Blau-Weiß-Selektion
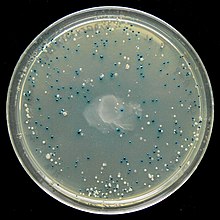
Die Blau-Weiß-Selektion ist eine Methode der Gentechnik zur Identifikation von bakteriellen Klonen, die ein Transgen enthalten.[1]
Prinzip
[Bearbeiten | Quelltext bearbeiten]
Das Ziel der Blau-Weiß-Selektion ist die Identifikation transgener Bakterien mit gewünschten Modifikationen. Zum einen werden diejenigen Bakterien identifiziert, die ein Plasmid (üblicherweise ein Klonierungsvektor) aufgenommen haben. Zum anderen wird durch die Blau-Weiß-Selektion die Frage beantwortet, ob das Plasmid erfolgreich modifiziert wurde. Für die Bestimmung Plasmid-enthaltender Bakterien tragen die Plasmide ein Gen, welches eine Antibiotikaresistenz vermittelt, die der verwendete Bakterienstamm nicht bereits selbst besitzt. Kultiviert man die Bakterien auf einem Kulturmedium mit dem zur Resistenz passenden Antibiotikum, so überleben dort idealerweise nur diejenigen Bakterien, die ein Plasmid aufgenommen haben.
Für die Blau-Weiß-Selektion werden bestimmte Plasmide als Vektoren verwendet, die an der Position zur Insertion des Transgens in das Plasmid (an der Multiple Cloning Site) das Gen für eine β-Galactosidase (lacZ-Gen) enthalten. Das Gen für die Galactosidase wird als Reportergen verwendet.
Durch Einfügung eines Transgens in die Multiple Cloning Site wird die Galactosidase inaktiviert. Dadurch enthalten nach einer Transformation der Plasmide die transgenen Organismen im Gegensatz zu den nicht transgenen Organismen keine funktionsfähige Galactosidase, sofern der zur Transformation verwendete Bakterienstamm kein weiteres Gen für eine Galactosidase enthält. Hierfür werden spezielle Mutantenstämme verwendet (z. B. E. coli Stämme JM109, DH5α und XL1-Blue), die eine Mutation enthalten (z. B. lacZΔM15, aus E. coli M15), welche die Expression einer funktionsfähigen Galactosidase unterdrückt. Die lacZΔM15-Mutante der Galactosidase besitzt eine Deletion der Aminosäuren 11 bis 41, wodurch die Galactosidase kein Homotetramer mehr bilden kann, welches die enzymatisch aktive Form der Galactosidase ist. Die lacZΔM15-Mutante der Galactosidase (synonym ω-Peptid) kann jedoch durch ein Protein der Aminosäuren 1 bis 59 der Galactosidase (α-Peptid) zu einer funktionsfähigen Form ergänzt werden, dessen Gen sich in diesem Fall auf dem Plasmid befindet.
Die Galactosidase ist eine Glycosidase und kann den gelben Farbstoff X-Gal in einen blauen unlöslichen Farbstoff (5,5'-Dibromo-4,4'-Dichloro-Indigo) und Galactose spalten. Das Gen lacZ der Galactosidase wird von einem Promotor kontrolliert, bei dem die Genexpression durch einen Induktor (z. B. Lactose, Allolactose, der stärkste bekannte Induktor ist IPTG) eingeleitet wird, da der Lac-Repressor LacI durch Bindung des Induktors das Gen zur Genexpression freigibt. Bei Verwendung von X-Gal im Kulturmedium entsteht etwa eine Stunde nach der Induktion durch die in den Bakterien enthaltene Galactosidase ein blauer Farbstoff. Bei den transgenen Organismen ist dagegen die Galactosidase inaktiviert, wodurch deren Kolonien ungefärbt bleiben und sie anhand ihrer fehlenden Färbung isoliert werden können. Da der Import von Lactose über die Lactosepermease (Gen lacY) durch Glucose gehemmt wird, sollte das Kulturmedium keine Glucose enthalten.
Nicht jede weiße Kolonie ist ein transgener Organismus (z. B. wegen LacZ-Inaktivierung durch DNA-Reparatur nicht ligierter Plasmide) und nicht jeder transgene Organismus enthält das gewünschte zu klonierende Gen, sondern andere DNA-Fragmente. Wenn zu wenig Antibiotikum oder zu alte Kulturmedien zur Selektion auf das Resistenzgen des Plasmids verwendet werden, wachsen zudem auch weiße Kolonien ohne Plasmid. Aus diesen Gründen erfolgt eine weitere Analyse, z. B. per Polymerasekettenreaktion (meistens Kolonie-Polymerasekettenreaktion) oder per DNA-Extraktion mit Restriktionsverdau, beide mit anschließender Agarose-Gelelektrophorese. X-Gal ist lichtempfindlich, weshalb der Farbstoff selbst und X-Gal-enthaltende Agarplatten im Dunklen aufbewahrt werden.
Geschichte
[Bearbeiten | Quelltext bearbeiten]Die Blau-Weiß-Selektion wurde zuerst in Komplementationsversuchen verwendet, d. h. durch Insertion eines zuvor entfernten DNA-Stücks sollte die Funktion der Galactosidase wiederhergestellt werden.[2][3][4] Die Blau-Weiß-Selektion zu Klonierungszwecken wurde erstmals in pUC-Plasmiden (z. B. pUC19) verwendet.[5]
Literatur
[Bearbeiten | Quelltext bearbeiten]- Cornel Mülhardt: Der Experimentator: Molekularbiologie/Genomics. Sechste Auflage. Spektrum Akademischer Verlag, Heidelberg 2008. ISBN 3-8274-2036-9.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ M. Koenen, U. Rüther, B. Müller-Hill: Immunoenzymatic detection of expressed gene fragments cloned in the lac Z gene of E. coli. In: The EMBO journal. Band 1, Nummer 4, 1982, ISSN 0261-4189, S. 509–512, PMID 6329687, PMC 553076 (freier Volltext).
- ↑ A. Ullmann, Francois Jacob, Jacques Monod: Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the beta-galactosidase structural gene of Escherichia coli. In: Journal of molecular biology. Band 24, Nummer 2, März 1967, ISSN 0022-2836, S. 339–343, PMID 5339877.
- ↑ K. E. Langley, M. R. Villarejo, A. V. Fowler, P. J. Zamenhof, I. Zabin: Molecular basis of beta-galactosidase alpha-complementation. In: Proceedings of the National Academy of Sciences. Band 72, Nummer 4, April 1975, ISSN 0027-8424, S. 1254–1257, PMID 1093175, PMC 432510 (freier Volltext).
- ↑ J. Messing, B. Gronenborn, B. Müller-Hill, P. Hans Hopschneider: Filamentous coliphage M13 as a cloning vehicle: insertion of a HindII fragment of the lac regulatory region in M13 replicative form in vitro. In: Proceedings of the National Academy of Sciences. Band 74, Nummer 9, September 1977, ISSN 0027-8424, S. 3642–3646, PMID 333444, PMC 431673 (freier Volltext).
- ↑ J. Vieira, J. Messing: The pUC plasmids, an M13mp7-derived system for insertion mutagenesis and sequencing with synthetic universal primers. In: Gene. Band 19, Nummer 3, Oktober 1982, ISSN 0378-1119, S. 259–268, PMID 6295879.