T-Zell-Rezeptor
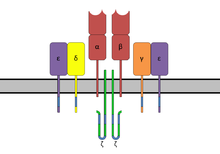
Der T-Zell-Rezeptor (englisch T cell receptor, TCR) ist ein Proteinkomplex, der auf der Oberfläche von T-Zellen verankert ist und für die Erkennung von Antigenen, die durch MHC-Moleküle präsentiert werden, zuständig ist. Er besitzt die besondere Eigenschaft, körperfremde von körpereigenen Antigenen zu unterscheiden und nur im ersten Fall die T-Zelle zu aktivieren. Die Bindung eines körperfremden zum Rezeptor passenden Antigens bewirkt über den TCR-Signalweg eine Veränderung der Genexpression. Diese führt zu Proliferation der Zelle, Sekretion von Zytokinen, Oberflächenexpression von Kostimulatoren und Differenzierung in Effektorzellen und Gedächtniszellen.
Struktur
[Bearbeiten | Quelltext bearbeiten]Der TCR ist strukturell einem Fab-Fragment der Antikörper sehr ähnlich, da seine Untereinheiten ebenfalls aus der Immunglobulin-Superfamilie stammen. Der TCR-Komplex besteht aus zwei Protein-Untereinheiten (zumeist α/β, in etwa 5 % der Zellen γ/δ), die wiederum jeweils aus einer konstanten Domäne (C) und einer variablen Domäne (V), einer Transmembrandomäne und einem kurzen C-terminalen cytoplasmatischen Bereich bestehen. Die N-terminalen Enden der Ketten, die zur C-Domäne gehören, durchdringen die Zellmembran bis in den cytoplasmatischen Raum und verankern den Rezeptor. Die beiden Untereinheiten sind extrazellulär über eine Disulfidbrücke in der konstanten Region miteinander verbunden. Das Homodimer der ζ-Ketten ist u.a für die Signalamplifikation und die Thymozyten-Selektion (Vorläuferzellen der T-Lymphozyten) bedeutend.[1]
Die variablen Domänen der α- und β-Untereinheit setzen sich aus V- und J-Segmenten bzw. für die β-Kette aus V-, D- und J-Segmenten zusammen, die je drei hypervariable (und an der β-Kette mit HV4 eine vierte, jedoch ohne Antigenkontakt) und somit Bindungs-entscheidende Bereiche (engl. complementarity determining regions, CDRs) bilden. Dabei interagiert die CDR 2 vornehmlich mit den α-Helices an der Peptidbindungsspalte der MHC-Klasse- I und II-Komplexe, während die CDR 1 der α-Kette an den N-Terminus und die CDR 1 der β-Kette an den C-Terminus des Antigens bindet und die beiden CDR3-Abschnitte den größten Anteil zur Bindung des MHC-präsentierten Antigens beitragen. Letztere weisen vor allem durch die kombinatorische Vielfalt bei der V(D)J-Rekombination eine größere Diversität auf. Die CDR4 interagiert zwar nicht mit Antigenen, jedoch sind Wechselwirkungen mit Superantigenen beschrieben.
Ob ein TCR ein Antigen binden kann oder nicht, ist ein sehr komplexer Prozess. Allgemein gilt das Schlüssel-Schloss-Prinzip. Wenn also die Struktur eines präsentierten Antigens im Zusammenhang mit dem präsentierenden MHC-Molekül an die α- und β-Kette des TCR passt, so kommt es zur Bindung. Computergestützte Molekül- und Bindungssimulationen sind ein Aufgabengebiet der Bioinformatik. Die Spezifität des TCR kann durch Epitopkartierung bestimmt werden.
Entstehung
[Bearbeiten | Quelltext bearbeiten]Neben der Struktur ist auch die Entstehung der T-Zell-Rezeptoren jener der Antikörper der B-Zellen ähnlich. Die beiden Untereinheiten entstehen durch genetische V(D)J-Rekombination (VJ in der α- oder γ-Kette, VDJ in der β- oder δ-Kette). Es kommt zu einer quasi zufälligen Anordnung der Gene, um eine möglichst große Diversität zu gewährleisten. Zusätzlich erfolgen Insertionen von N- oder P-Nukleotiden. Diese ist der Grundbaustein der adaptiven Immunantwort bei T-Zellen. Die T-Zell-Klone mit jeweils einzigartigem T-Zell-Rezeptor unterliegen im Thymus positiver und negativer Selektion, um T-Zellen mit nicht-funktionellen oder selbst-reaktiven Produkten der Rekombination auszusortieren.
Antigendiskriminierung
[Bearbeiten | Quelltext bearbeiten]Eine einzigartige Eigenschaft des T Zell Rezeptors ist die Fähigkeit, körperfremde Antigene von Krankheitserregern sowie Krebszellen von körpereigenen Antigenen zu unterscheiden.[2] Antigenpräsentierende Zellen unterscheiden nicht zwischen körperfremden und körpereigenen Zellen. Sie präsentieren neben wenigen körperfremden Antigenen eine Vielzahl von körpereigenen Antigenen auf ihrer Zelloberfläche.[3] Der T-Zell-Rezeptor ist in der Lage, durch die wenigen körperfremden Antigene, an die er mit hoher Affinität bindet, aktiviert zu werden, jedoch nicht von der Vielzahl an körpereigenen Antigenen, an die er nur mit geringer Affinität bindet. Dieses Phänomen wird als Antigendiskriminierung bezeichnet. Der T-Zell-Rezeptor ist sowohl sehr spezifisch als auch sehr sensitiv, um diese Antigendiskriminierung zu ermöglichen.[2] Die Aktivierung der T-Zellen korreliert mit der Affinität des T-Zell-Rezeptors zum Antigen-MHC-Komplex.[4] Der TCR ist so spezifisch, sodass er selbst die Veränderung einer einzigen Aminosäure im Antigen erkennen kann. Dieser Verlust in Affinität kann selbst durch höhere Antigenkonzentrationen nicht ausgeglichen werden.[5] Trotz der großen Spezifität besitzt der Rezeptor eine sehr hohe Sensitivität. Die Interaktion mit einem einzigen Antigen-MHC Komplex ist ausreichend, um die T Zelle zu aktivieren.[6]
Der Mechanismus dieser sich eigentlich gegenseitig ausschließenden Eigenschaften der Spezifität und Sensitivität ist noch nicht vollständig verstanden. Stöchiometrische Untersuchungen an TCRs und den assoziierten CD3-Untereinheiten auf den Oberflächen lebender T-Zellen zeigen, dass monomere TCR-CD3 Komplexe der Erkennung der antigenen pMHCs zugrunde liegen.[7]
Signaltransduktion
[Bearbeiten | Quelltext bearbeiten]Da der T-Zell-Rezeptor selbst nur einen sehr kurzen cytoplasmatischen Bereich besitzt, bildet er einen Komplex mit CD3-Adapterproteinen, die in ihrer langen cytoplasmatischen Aminosäurenkette Signaltransduktionsmotive, sog. Immunglobulin-Tyrosin-Aktivierungsmotive (ITAMs), besitzen.[8] CD3 besteht in Säugern aus der γ-, δ- und ε-Kette sowie zusätzlich aus Komplexen von ζ2- oder ζ/η-Ketten. Der Gesamtkomplex wird als T-Zell-Rezeptor-Komplex bezeichnet. Nach der Bindung eines Antigen-MHC-Komplexes zur Aktivierung des TCR und Bindung des Co-Rezeptors CD4 oder CD8 an MHC phosphorylieren die Tyrosinekinasen Lck und Fyn Tyrosinreste in den ITAMs. Die Tyrosinphosphatase CD45 kann die Phosphoylierungen wieder entfernen und somit das TCR-Signal abschalten. Gleichzeitig kann CD45 aber auch Lck und Fyn durch Dephosphorylierung aktivieren. Die phosphorylierten ITAMs dienen als Bindestellen für ZAP-70, welches durch diese Bindung in die Nachbarschaft von Lck gebracht wird.[9] Lck phosphoryliert ZAP-70, welches nun das Adapterprotein LAT an mehreren Stellen phosphorylieren kann. LAT ist ein Gerüstprotein, es hat selbst eine katalytische Aktivität, dient aber als Bindepartner für viele weitere Signalproteine. So entsteht ein großer Proteinkomplex, der die nachstehenden Signalwege aktivieren kann. Unter anderem werden verschiedene MAP-Kinase Wege aktiviert, die zur Aktivierung des Transkriptionsfaktors AP-1 führen. Des Weiteren kann die Aktivierung der Phospholipase C-γ (PLC-γ) durch Bindung an LAT erfolgen. PLC-γ produziert die Second Messenger Diacylglycerol (DAG) und Inositoltrisphosphat (IP3), welche die Transkriptionsfaktoren NF-κB, beziehungsweise NFAT via Calciumeinstrom in die Zelle aktiviert.[10]
Co-Rezeptoren
[Bearbeiten | Quelltext bearbeiten]Das durch die TCR-Antigen-Bindung entstehende Signal wird durch die simultane Bindung an Co-Rezeptoren verstärkt. Der CD4- und CD8-Rezeptor binden an das MHC-Molekül nach dem der TCR an das Antigen bindet. Lck, welches an die cytoplasmatische Domaine der Co-Rezeptoren bindet, wird so in die Nähe des TCR gebracht. Der CD4-Rezeptor bindet ausschließlich MHC II, während der CD8-Rezeptor spezifisch für MHC I ist. CD4 und CD8 bestimmen die Differenzierung in T-Helferzelle bzw. cytotoxischen T-Zelle während der Entwicklung im Thymus.[10] Die Bindung von den Co-Rezeptoren hat auch Auswirkung auf die Spezifität und Sensitivität des T-Zell-Rezeptors.[11]
Bindung von Co-Stimulierenden Rezeptoren und ihre Liganden auf Antigen-präsentierenden Zellen verstärken die Aktivierung der T Zelle. Ein Beispiel ist der CD28 Rezeptor auf T-Zellen, welcher die Proteine der B7-Familie (CD80 und CD86) bindet. Antigen-präsentierende Zellen exprimieren die B7-Proteine auf ihrer Oberfläche, wenn sie, als Teil des angeborenen Immunsystems, eine Infektion mit einem Krankheitserreger erkannt haben. Die Co-Stimulierenden Rezeptoren liefern ein zweites Signal zusätzlich zum TCR, sozusagen ein Signal, was Gefahr signalisiert, und nur wenn dieses zusätzlich zur Erkennung eines Antigens durch den TCR vorhanden ist, kann die T-Zelle aktiviert werden. Dieses System sorgt dafür, dass T-Zellen, die aus Versehen ein körpereigenes Antigen erkennen, nicht aktiviert werden.[12] Manche Co-Stimulierenden Rezeptoren sind allerdings nicht unbedingt notwendig, sondern verstärken das TCR-Signal nur.[12]
Des Weiteren gibt es Co-inhibitorische Rezeptoren, z. B. CTLA-4 und PD-1, welche das TCR-Signal vermindern oder blockieren. Sie spielen eine wichtige Rolle zur Regulierung und Abschalten der Immunreaktion, wenn der Krankheitserreger erfolgreich bekämpft wurde.[12]
TCR-Immuntherapien
[Bearbeiten | Quelltext bearbeiten]Ein immuntherapeutischer Ansatz versucht, die bestehende Toleranz gegenüber den Krebszellen und die Tumor-induzierte Unterdrückung einer Immunantwort im Patienten zu überwinden. Hierfür werden die T‑Zellen des Patienten außerhalb des Körpers aktiviert, mit Tumor-spezifischen TCRs gentechnisch modifiziert und anschließend vermehrt. Damit kann eine große Anzahl an spezifischen T‑Zellen, die den Tumor bekämpfen können, für Patienten innerhalb kurzer Zeit verfügbar gemacht werden. Im Februar 2022 gaben BioNTech und Medigene, ein Immunonkologie-Unternehmen mit klinischen Projekten fokussiert auf die Entwicklung T-Zell-gerichteter Krebstherapien, den Abschluss einer Forschungskollaboration zur Entwicklung von T-Zell-Rezeptor („TCR“)-basierten Immuntherapien zur Behandlung von Krebs bekannt.[13]
Literatur
[Bearbeiten | Quelltext bearbeiten]- Janeway et al.: Immunobiology. 6. Auflage. ISBN 0-8153-4101-6. Die 5. englische Ausgabe ist online auf den Seiten des NCBI-Bookshelf verfügbar, ncbi.nlm.nih.gov.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Jennifer McDowall: Protein Of The Month: T-cell receptors. ebi.ac.uk, Interpro (englisch).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ E. W. Shores, P. E. Love: TCR zeta chain in T cell development and selection. In: Current Opinion in Immunology. Band 9, Nr. 3, Juni 1997, ISSN 0952-7915, S. 380–389, doi:10.1016/s0952-7915(97)80085-4, PMID 9203416.
- ↑ a b Feinerman O, Germain RN, Altan-Bonnet G: Quantitative challenges in understanding ligand discrimination by alphabeta T cells. In: Molecular Immunology. 45. Jahrgang, Nr. 3, Februar 2008, S. 619–31, doi:10.1016/j.molimm.2007.03.028, PMID 17825415, PMC 2131735 (freier Volltext).
- ↑ Blum JS, Wearsch PA, Cresswell P: Pathways of antigen processing. In: Annual Review of Immunology. 31. Jahrgang, 2013, S. 443–73, doi:10.1146/annurev-immunol-032712-095910, PMID 23298205, PMC 4026165 (freier Volltext).
- ↑ Dushek O, Aleksic M, Wheeler RJ, Zhang H, Cordoba SP, Peng YC, Chen JL, Cerundolo V, Dong T, Coombs D, van der Merwe PA: Antigen potency and maximal efficacy reveal a mechanism of efficient T cell activation. In: Science Signaling. 4. Jahrgang, Nr. 176, Juni 2011, S. ra39, doi:10.1126/scisignal.2001430, PMID 21653229, PMC 4143974 (freier Volltext).
- ↑ Altan-Bonnet G, Germain RN: Modeling T cell antigen discrimination based on feedback control of digital ERK responses. In: PLoS Biology. 3. Jahrgang, Nr. 11, November 2005, S. e356, doi:10.1371/journal.pbio.0030356, PMID 16231973, PMC 1262625 (freier Volltext).
- ↑ Huang J, Brameshuber M, Zeng X, Xie J, Li QJ, Chien YH, Valitutti S, Davis MM: A single peptide-major histocompatibility complex ligand triggers digital cytokine secretion in CD4(+) T cells. In: Immunity. 39. Jahrgang, Nr. 5, November 2013, S. 846–57, doi:10.1016/j.immuni.2013.08.036, PMID 24120362, PMC 3846396 (freier Volltext).
- ↑ Mario Brameshuber, Florian Kellner, Benedikt K. Rossboth, Haisen Ta, Alge Kevin, Eva Sevcsik, Janett Göhring, Markus Axmann, Florian Baumgart, Nicholas R. J. Gascoigne, Simon J. Davis, Hannes Stockinger, Gerhard J. Schütz, Johannes B. Huppa: Monomeric TCRs drive T cell antigen recognition. In: Nat. Immunol. 16. April 2018, doi:10.1038/s41590-018-0092-4.
- ↑ Call ME, Pyrdol J, Wiedmann M, Wucherpfennig KW: The organizing principle in the formation of the T cell receptor-CD3 complex. In: Cell. 111. Jahrgang, Nr. 7, Dezember 2002, S. 967–79, doi:10.1016/s0092-8674(02)01194-7, PMID 12507424, PMC 3420808 (freier Volltext).
- ↑ Clare L. Abram, Clifford A. Lowell: The Expanding Role for ITAM-Based Signaling Pathways in Immune Cells. In: Science Signalling. 2007. Jahrgang, Nr. 377, 13. März 2007, S. re2.
- ↑ a b Janeway's immunobiology. Ninthition, ISBN 0-8153-4551-8.
- ↑ JD Stone, AS Chervin, DM Kranz: T-cell receptor binding affinities and kinetics: impact on T-cell activity and specificity. In: Immunology. 126. Jahrgang, Nr. 2, Februar 2009, S. 165-76, doi:10.1111/j.1365-2567.2008.03015.x, PMID 19125887.
- ↑ a b c Lieping Chen, Dallas B. Flies: Molecular mechanisms of T cell co-stimulation and co-inhibition. In: Nature Reviews Immunology. 13. Jahrgang, Nr. 4, 8. März 2013, S. 227–242, doi:10.1038/nri3405.
- ↑ BioNTech und Medigene gehen globale Kollaboration zur Entwicklung von T-Zell-Rezeptor-Immuntherapien gegen Krebs ein, PM BioNTech vom 21. Februar 2022, abgerufen am 24. Juni 2022