JC-Virus
| JC-Virus | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
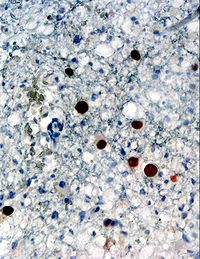
Immunhistochemischer Nachweis von JC-Virusprotein (braun angefärbt) in einer Hirnbiopsie | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| JC polyomavirus | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| JCPyV | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Das JC-Virus (JC-Polyomavirus, John Cunningham virus; Kürzel: JCPyV, JCV) gehört als Typusstamm der Virusspezies Betapolyomavirus secuhominis (Humanes Polyomavirus 2, HPyV-2) zusammen mit dem SV-40-Virus (SV40-Virus, Spezies Betapolyomavirus macacae) und dem BK-Virus (Spezies Betapolyomavirus hominis, Humanes Polyomavirus 1, HPyV-1) zur Familie der Polyomaviridae und darin zur Gattung Betapolyomavirus.[2][3][4] Es gilt als opportunistischer Erreger einer Gehirnerkrankung mit Entmarkungsherden, der sogenannten Progressiven Multifokalen Leukenzephalopathie (PML).
Namensgebung
[Bearbeiten | Quelltext bearbeiten]Die Bezeichnung „JC-Virus“ setzt sich aus den Initialen von John Cunningham, dem Patienten, bei dem das Virus 1971 erstmals isoliert wurde, zusammen.[5]
Übertragungsweg
[Bearbeiten | Quelltext bearbeiten]Lange Zeit wurde davon ausgegangen, dass die Übertragung des Virus über den Respirationstrakt erfolgt, da virale DNA in Lymphozyten der Tonsillen nachgewiesen werden konnte. Inzwischen gibt es auch Hinweise auf einen fäkal-oralen Übertragungsweg mit Virusnachweis im Stuhl.
Infektion
[Bearbeiten | Quelltext bearbeiten]Die Infektion erfolgt in der Regel im Kindesalter; etwa 85 % der Erwachsenen weltweit zeigen Antikörper gegen das JC-Virus, die auf eine durchgemachte Erkrankung hinweisen. In der Regel fehlen klinische Symptome, das Virusgenom verbleibt jedoch latent in Epithelzellen der Niere, fraglich auch im Knochenmark. Bei Schwäche des Immunsystems kann das Virus reaktiviert und hämatogen gestreut werden und in Oligodendrozyten des ZNS einwandern, wo sich dann eine PML entwickeln kann.
Der Nachweis der verschiedenen Subtypen des Virus wurde als genetischer Marker für Migration und Evolution der menschlichen Population verwendet, jedoch eignet er sich dazu nicht.[6]
Aufbau
[Bearbeiten | Quelltext bearbeiten]Das ikosaedrische Kapsid hat einen Durchmesser von etwa 45 nm und ist aus 72 Kapsomeren, sog. VP-1-Pentameren, aufgebaut. Die anderen Hüllproteine VP-2 und VP-3 sind nur in geringer Anzahl enthalten.[7]
Das zirkuläre doppelsträngige Genom des JC-Virus besteht aus 5130 Basenpaaren (bp) und wird in drei Abschnitte unterteilt. Zum einen, eine nichtcodierende Region mit Replikationsursprung, eine Region für das große und kleine T-Antigen, sowie eine Region für die Hüllproteine VP-1, VP-2 und VP-3 sowie das dazugehörige Agnoprotein. Durch unterschiedliche Arrangements der nichtcodierenden Region entstehen unterschiedliche JC-Virusvarianten. Am häufigsten findet man in gesunden Individuen den CY-Stamm mit einer 98 bp langen Sequenz bestehend aus zwei 23 und 66 bp Einsatzstücken, während in PML-Patienten der Mad-1 Stamm aus zwei 98 bp Tandem-Repeats und TATA-Boxen besteht. Insbesondere der Mad-4-Stamm besitzt onkogenes Potential, da die zweite TATA-Box hier fehlt.
Im Tiermodell werden durch Infektion mit dem JC-Virus, Stamm Mad-4, zahlreiche Gehirntumoren, insbesondere Astrozytome, Medulloblastome und PNET beobachtet. Auch wird gelegentlich in Operationsproben menschlicher ZNS-Tumore JC-Virus-DNA nachgewiesen. Insbesondere das 80-kDa-Protein des großen T-Antigens kann bestimmte Schlüsselproteine für die Entstehung von Tumoren (p53 und pRb) binden und inaktivieren.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- B. Vagner Ramirez, V. Castano Palacio: JC virus: A brief review. In: World J. Neurosci. 3, 2013, 126–130, doi:10.4236/wjns.2013.32015
- Hirntumor durch Viren?, Spektrum
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c d e ICTV: ICTV Taxonomy history: Human polyomavirus 1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ José Carlos Mann Prado, Telma Alves Monezi, Aline Teixeira Amorim, Vanesca Lino, Andressa Paladino, Enrique Boccardo: Human polyomaviruses and cancer: an overview. In: Clinics (Sao Paulo), 73(Suppl 1), S. e558s, doi:10.6061/clinics/2018/e558s, PMC 6157077 (freier Volltext), PMID 30328951 (online 26. September 2018), Fig. 1
- ↑ Ugo Moens, Sébastien Calvignac-Spencer, Chris Lauber, Torbjörn Ramqvist, Mariet C. W. Feltkamp, Matthew D. Daugherty, Ernst J. Verschoor, Bernhard Ehlers: ICTV Virus Taxonomy Profile: Polyomaviridae. In: J Gen Virol., 98(6), S. 1159–1160, doi:10.1099/jgv.0.000839, PMC 5656788 (freier Volltext), PMID 28640744, 22. Juni 2017
- ↑ dsDNA Viruses > Polyomaviridae. In: ICTV Report, Juni 2017, überarbeitet im Juli 2018, Tabelle 2A
- ↑ B. L. Padgett, D. L. Walker, G. M. Zu Rhein u. a.: Cultivation of papova-like virus from human brain with progressive multifocal leucoencephalopathy. In: Lancet, 1, 1971, S. 1257–1260. PMID 4104715
- ↑ Laura A. Shackelton, Andrew Rambaut, Oliver G. Pybus, Edward C. Holmes: JC Virus Evolution and Its Association with Human Populations. In: Journal of Virology. Band 80, Nr. 20, 15. Oktober 2006, ISSN 0022-538X, S. 9928–9933, doi:10.1128/JVI.00441-06 (asm.org [abgerufen am 18. April 2024]).
- ↑ H. Petry, C. Goldmann, O. Ast, W. Lüke: The use of virus-like particles for gene transfer. In: Curr Opin Mol Ther. 5, 2003, S. 524–528. PMID 14601522