Polymerase-Kettenreaktion
Als Polymerase-Kettenreaktion (englisch polymerase chain reaction, PCR) bezeichnet man Methoden zur in vitro-Vervielfältigung von Erbsubstanz (Desoxyribonukleinsäure). Dazu werden, je nach Methode, verschiedene Formen des Enzyms DNA-Polymerase verwendet. Die Bezeichnung Kettenreaktion bedeutet in diesem Zusammenhang, dass die Produkte vorheriger Zyklen als Ausgangsstoffe für die nächsten Zyklen dienen und somit eine exponentielle Vervielfältigung ermöglichen.
Bei der klassischen PCR werden diese Zyklen über ein Temperaturprogramm gesteuert. Bei der Weiterentwicklung isotherme DNA-Amplifikation erfolgt diese Vervielfältigung kontinuierlich bei konstanter Temperatur.
Die PCR wird in biologischen und medizinischen Laboratorien zum Beispiel für die Erkennung von Erbkrankheiten und Virusinfektionen, für das Erstellen und Überprüfen genetischer Fingerabdrücke, für das Klonieren von Genen und für Abstammungsgutachten verwendet. Sie ist die empfindlichste und zuverlässigste Labor-Methode des direkten Erregernachweises.
Entwickelt wurde die Methode durch den Biochemiker Kary Mullis im Jahr 1985[1][2]. 1993 wurde ihm dafür der Nobelpreis für Chemie verliehen. Die PCR zählt heute zu den wichtigsten Methoden der modernen Molekularbiologie, und viele wissenschaftliche Fortschritte auf diesem Gebiet (z. B. im Rahmen des Humangenomprojekts) wären ohne diese Methode nicht möglich gewesen.
Geschichte
[Bearbeiten | Quelltext bearbeiten]


Anfang der 1970er Jahre kam der norwegische Postdoc Kjell Kleppe im Labor von Nobelpreisträger Har Gobind Khorana auf den Gedanken, DNA durch zwei flankierende Primer zu vervielfältigen, jedoch geriet die Idee in Vergessenheit.[3] Die Polymerase-Kettenreaktion selbst wurde 1983 von Kary Mullis erneut erfunden. Seine Absicht war es, ein neuartiges DNA-Syntheseverfahren zu entwickeln, das DNA durch wiederholte Verdopplung in mehreren Zyklen mittels eines Enzyms namens DNA-Polymerase künstlich vervielfältigt. Sieben Jahre nach der Veröffentlichung seiner Idee wurde Mullis hierfür 1993 der Nobelpreis für Chemie verliehen.[4]
DNA-Polymerase kommt in allen Lebewesen vor und verdoppelt während der Replikation die DNA vor der Zellteilung. Dazu bindet sie sich an einen einzelnen DNA-Strang und synthetisiert mittels eines kurzen komplementären Oligonukleotids (Primer) einen dazu komplementären Strang. Bereits in Mullis’ ursprünglichem PCR-Versuch wurde das Enzym in vitro verwendet. Die doppelsträngige DNA wurde zunächst durch Erhitzen auf 96 °C in zwei Einzelstränge getrennt. Bei dieser Temperatur wurde die dabei zunächst verwendete DNA-Polymerase I von E. coli zerstört und musste daher nach jedem Erhitzen erneut zugegeben werden. Diese Folge von Arbeitsschritten musste mehrere dutzendmal in Folge wiederholt werden, um eine ausreichende Amplifikation zu erreichen. Mullis’ ursprüngliches Verfahren war daher sehr ineffizient, da es viel Zeit, große Mengen DNA-Polymerase und ständige Aufmerksamkeit erforderte.
Eine entscheidende Verbesserung der PCR-Technologie brachte die Verwendung von thermostabilen DNA-Polymerasen, das heißt von Enzymen, die auch bei Temperaturen von annähernd 100 °C ihre Polymerase-Aktivität behalten und nicht denaturieren. Eine der ersten thermostabilen DNA-Polymerasen wurde aus dem in heißen Quellen lebenden thermophilen Bakterium Thermus aquaticus gewonnen und Taq-Polymerase genannt. Durch die Verwendung thermostabiler DNA-Polymerasen bestand keine Notwendigkeit mehr, ständig neue Polymerase zuzugeben, und der ganze PCR-Prozess konnte erheblich vereinfacht und automatisiert werden.
Mullis arbeitete zu dieser Zeit für die kalifornische Biotech-Firma Cetus und wurde mit einer Prämie von 10.000 US-Dollar abgefunden. Diese meldete die PCR-Methode zum Patent an.[2] Jahre später verkaufte Cetus dann die Rechte an der PCR-Methode mitsamt dem Patent für die von ihm verwendete DNA-Polymerase Taq an die Firma Roche für 300 Millionen Dollar. Das Enzym Taq wurde jedoch bereits 1980 von dem russischen Forscher Kaledin beschrieben. Aus diesem Grund wurde nach jahrelangem Rechtsstreit der Firma Roche das Patent für Taq inzwischen entzogen. Die US-Patente für die PCR-Technologie selbst liefen im März 2005 aus.
Die Taq-Polymerase erfährt nach wie vor breite Anwendung. Ihr Nachteil liegt darin, dass sie manchmal Fehler beim Kopieren der DNA produziert, was zu Mutationen in der DNA-Sequenz führt. Polymerasen wie Pwo und Pfu, die aus Archaea gewonnen werden, haben einen Korrekturmechanismus, der die Anzahl der Mutationen in der kopierten DNA erheblich senkt.
Prinzip
[Bearbeiten | Quelltext bearbeiten]PCR wird eingesetzt, um einen kurzen, genau definierten Teil eines DNA-Strangs zu vervielfältigen. Der zu vervielfältigende Bereich der DNA wird auch als Amplicon bezeichnet, die bei der PCR entstehenden Produkte als Amplifikate. Dabei kann es sich um ein Gen oder auch nur um einen Teil eines Gens handeln oder auch um nichtcodierende DNA-Sequenzen. Im Gegensatz zu lebenden Organismen kann der PCR-Prozess nur relativ kurze DNA-Abschnitte kopieren. Bei einer Standard-PCR können dies bis zu etwa dreitausend Basenpaare (3 kbp) lange DNA-Fragmente sein. Mit Hilfe bestimmter Polymerasen-Gemische, weiterer Additive in der PCR und optimalen Bedingungen können sogar Fragmente mit einer Länge von über 20–40 kbp vervielfältigt werden, was immer noch sehr viel kürzer ist als die chromosomale DNA einer eukaryotischen Zelle. Eine menschliche Zelle enthält beispielsweise etwa drei Milliarden Basenpaare pro haploidem Genom.[5]
Komponenten und Reagenzien
[Bearbeiten | Quelltext bearbeiten]

Eine PCR benötigt mehrere grundlegende Komponenten. Diese sind:
- Die Original-DNA, die den zu vervielfältigenden Abschnitt enthält (Template)
- Zwei Primer, um auf den beiden Einzelsträngen der DNA jeweils den Startpunkt der DNA-Synthese festzulegen, wodurch der zu vervielfältigende Bereich von beiden Seiten begrenzt wird
- DNA-Polymerase, die bei hohen Temperaturen nicht zerstört wird, um den festgelegten Abschnitt zu replizieren (kopieren) (z. B. Taq-Polymerase)
- Desoxyribonukleosidtriphosphate, die Bausteine für den von der DNA-Polymerase synthetisierten DNA-Strang
- Mg2+ -Ionen, für die Funktion der Polymerase essentiell, stabilisieren die Anlagerung der Primer und bilden lösliche Komplexe mit den Desoxyribonucleosidtriphosphaten
- Pufferlösungen, die eine für die DNA-Polymerase geeignete chemische Umgebung sicherstellen
Die Reaktion wird üblicherweise in Volumina von 10–200 µl in kleinen Reaktionsgefäßen (200–500 µl) in einem Thermocycler durchgeführt. Diese Maschine erhitzt und kühlt die in ihr befindlichen Reaktionsgefäße präzise auf die Temperatur, die für den jeweiligen Schritt benötigt wird. Etwaige Kondensatbildung im Deckel des Gefäßes wird durch einen beheizbaren Gerätedeckel (über 100 °C) verhindert. Die Thermocycler der ersten Generation besaßen noch keinen beheizten Deckel, weshalb bei diesen Geräten zur Vermeidung einer Verdampfung von Wasser während der PCR die Reaktionsansätze mit Mineralöl überschichtet wurden. Als Reaktionsgefäß können, je nach Probeneinsatz bzw. Heizblock des Thermocyclers, neben einzelnen 200-µl-Reaktionsgefäßen auch acht zusammenhängende 200-µl-Reaktionsgefäße oder PCR-Platten für bis zu 96 Ansätze mit 200 µl oder auch 384 Ansätze zu je 50 µl verwendet werden. Die Platten werden entweder mit einer Gummiabdeckung oder einer selbstklebenden Klarsichtfolie verschlossen.
Beispiel
[Bearbeiten | Quelltext bearbeiten]Als allgemeines Beispiel sei hier eine typische Zusammensetzung einer PCR-Reaktion wiedergegeben. Viele Beispiele für die verschiedensten Variationen der PCR finden sich in der wissenschaftlichen Literatur in verschiedensten Kombinationen:
| 1,0 µl DNA-Lösung (100 ng/µl) |
| 2,0 µl Primer (i. d. R. zwei, jeweils 1 µl) (10 pmol/µl) |
| 4,0 µl 10 mmol Desoxy-Nukleotide (dATP, dGTP, dCTP, dTTP), je dNTP 1 µl |
| 5,0 µl 10-fach konzentrierte Polymerase-Pufferlösung |
| 37,0 µl H2O |
| 1,0 µl Pfu-, Taq- oder andere thermostabile DNA-Polymerase (1–5 U/µl) |
|
|
| 50,0 µl Gesamtvolumen |
Ablauf
[Bearbeiten | Quelltext bearbeiten]Der PCR-Prozess besteht aus etwa 20–50 Zyklen, die in einem Thermocycler durchgeführt werden. Die folgenden Angaben sind als Richtwerte gedacht. Meist muss eine PCR auf die spezifische Reaktion hin optimiert werden. Jeder Zyklus besteht aus drei Schritten (siehe Abbildung unterhalb):
- Denaturierung (Melting, Schmelzen): Zunächst wird die doppelsträngige DNA auf 94–96 °C erhitzt, um die Stränge zu trennen. Die Wasserstoffbrückenbindungen, die die beiden DNA-Stränge zusammenhalten, werden aufgebrochen. Im ersten Zyklus wird die DNA oft für längere Zeit erhitzt (Initialisierung), um sicherzustellen, dass sich sowohl die Ausgangs-DNA als auch die Primer vollständig voneinander getrennt haben und nur noch Einzelstränge vorliegen. Manche (sogenannte Hot-Start-) Polymerasen müssen durch eine noch längere anfängliche Erhitzungsphase (bis zu 15 Minuten) aktiviert werden.
- Primerhybridisierung (primer annealing): In diesem Schritt wird die Temperatur abgesenkt und ca. 30 Sekunden lang auf einem Wert gehalten, der eine spezifische Anlagerung der Primer an die DNA erlaubt. Die genaue Temperatur wird hierbei durch die Länge und die Sequenz der Primer bestimmt (bzw. der passenden Nukleotide im Primer, wenn durch diesen Mutationen eingeführt werden sollen = site-directed mutagenesis). Wird die Temperatur zu niedrig gewählt, können sich die Primer unter Umständen auch an nicht hundertprozentig komplementären Sequenzen anlagern und so zu unspezifischen Produkten („Geisterbanden“) führen. Wird die Temperatur zu hoch gewählt, ist die thermische Bewegung der Primer unter Umständen so groß, dass sie sich nicht richtig anheften können, so dass es zu gar keiner oder nur ineffizienter Produktbildung kommt. Die Temperatur, welche die beiden oben genannten Effekte weitgehend ausschließt, liegt normalerweise 5–10 °C unter dem Schmelzpunkt der Primersequenzen; dies entspricht meist einer Temperatur von 55 bis 65 °C.
- Elongation (Extension, Polymerisation, Verlängerung, Amplifikation): Schließlich füllt die DNA-Polymerase die fehlenden Stränge mit freien Nukleotiden auf. Sie beginnt am 3'-Ende des angelagerten Primers und folgt dann dem DNA-Strang. Der Primer wird nicht wieder abgelöst, er bildet den Anfang des neuen Einzelstrangs. Die Temperatur hängt vom Arbeitsoptimum der verwendeten DNA-Polymerase ab (68–72 °C). Bei diesen Temperaturen können jedoch nur ganz bestimmte Enzyme arbeiten. Oft wird hier die Taq-Polymerase genutzt. Dieser Schritt dauert etwa 30 Sekunden je 500 Basenpaare, variiert aber in Abhängigkeit von der verwendeten DNA-Polymerase. Die PCR-Produkte sind anschließend viele Tage auch bei Raumtemperatur stabil, sodass eine Weiterverarbeitung nicht sofort erfolgen muss. In vielen Laboren hat es sich etabliert, die Proben im Thermocycler nach Ende der PCR auf 4–8 °C herunter zu kühlen. Viele Hersteller raten davon jedoch ab, da aufgrund von Kondensation im Metallblock die Lebenszeit eines Cyclers mit Peltier-Element stark reduziert wird.[6]

- (A) Die Ausgangs-DNA liegt zunächst als Doppelstrang vor (Pfeilrichtung: 5'→3').
- (B,C) Nach der Denaturierung sind die Einzelstränge getrennt und die Primer können binden.
- (C,D) Die Polymerase produziert den Gegenstrang, indem sie die Primer 5'→3' (in Pfeilrichtung) verlängert. Die Produkte sind jeweils noch an einem Ende zu lang. Dies ist damit zu erklären, dass lediglich ein Startpunkt (Primer), nicht aber ein Endpunkt exakt festgelegt ist.
- (E,F) Im nächsten Zyklus entstehen erstmals PCR Produkte in der richtigen Länge – allerdings sind die Gegenstränge jeweils noch zu lang.
- (G,H) Im dritten Zyklus entsteht erstmals das PCR Produkt als Doppelstrang in der richtigen Länge (die anderen Produkte sind in H nicht dargestellt).
- (H,I,J) In den folgenden Zyklen vermehren sich die gewünschten Produkte exponentiell (da sie selbst als Matrize für weitere Strangsynthesen dienen), während die ungewünschten langen Produkte (siehe Produkte des ersten Zyklus) nur linear ansteigen (nur eingesetzte DNA dient als Matrix).
Dies ist der theoretische Idealfall, in der Praxis fallen aber zudem in geringem Maße auch kürzere Fragmente als die gewünschte Ziel-DNA an. Diese kurzen Fragmente häufen sich vor allem in den späten Zyklen an und können durch Fehlpaarung der Primer auch zu falschen PCR Produkten werden. Daher werden bei PCR Reaktionen meist nur etwa 30 Zyklen durchlaufen, damit vorwiegend DNA der gewünschten Länge und Sequenz produziert wird.
Aufreinigung von PCR-Produkten
[Bearbeiten | Quelltext bearbeiten]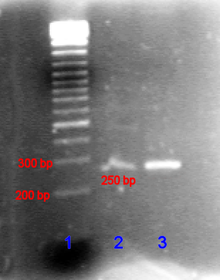
Ein PCR-Produkt kann durch Agarose-Gelelektrophorese anhand seiner Größe identifiziert werden. (Die Agarose-Gelelektrophorese ist ein Verfahren, bei der DNA in ein Agarose-Gel eingebracht wird und anschließend eine Spannung angelegt wird. Dann bewegen sich die kürzeren DNA-Stränge schneller als die längeren auf den Pluspol zu.) Die Länge des PCR-Produkts kann durch einen Vergleich mit einer DNA-Leiter, die DNA-Fragmente bekannter Größe enthält und parallel zur Probe im Gel mitläuft, bestimmt werden. Soll die PCR vor allem als quantitativer Nachweis dienen, empfiehlt sich die Real Time PCR oder die Digital PCR.
PCR-Optimierung
[Bearbeiten | Quelltext bearbeiten]Verschiedene Methoden können eingesetzt werden, um die Synthesemengen zu steigern oder Inhibitoren der PCR zu beseitigen.
Varianten
[Bearbeiten | Quelltext bearbeiten]Die klassische PCR ist durch zahlreiche Variationen erweitert und verbessert worden. Dadurch können verschiedene Aufgaben spezifisch angegangen werden. Alternativ zur PCR können auch verschiedene Methoden der isothermalen DNA-Amplifikation oder Ligase-Kettenreaktion verwendet werden.
- Agglutinations-PCR: Methode zur Bestimmung der Menge von Antikörpern. Es werden Antigene durch Antikörper isoliert und dann vermehrt. Die genaue Antikörpermenge muss aber schon vorher vorliegen, z. B. im Neutralisationshemmtest.
- Digital PCR (dPCR): Bei der digital PCR (dPCR) wird die DNA verdünnt und auf eine große Anzahl an Femtoliter-Reaktionsgefäßen verteilt. Pro Reaktionsgefäß entsteht entweder DNA oder nicht. Aufgenommen wird ein Digitalsignal. Durch Auszählen einer großen Anzahl an Reaktionsgefäßen kann der Anteil erfolgter Reaktionen zur Mengenbestimmung verwendet werden.
- Immun-PCR: Methode zur Erkennung von Antigenen.
- Immunoquantitative Echtzeit-PCR (irt-PCR): Manchmal müssen selbst geringe Mengen an Pathogenen wirksam erkannt werden, da sie auch einzeln für den Menschen gefährlich werden können. Die Detektionsschwelle vieler immunologischer Methoden (z. B. ELISA) kann für diese Fälle unzureichend sein, so dass man hier auf die immunoquantitative Echtzeit-PCR (immunoquantitative real-time PCR) zurückgreift.[7] Hierbei kombiniert man die hohe Spezifität von Antikörpern mit einer qPCR. Wie beim klassischen ELISA nutzt man zwei Antikörper. Der erste wird an einer Mikrotiterplatte fixiert und erkennt das gesuchte Antigen. Daran bindet dann der zweite Antikörper. Im herkömmlichen ELISA wird der immunologische Komplex aus erstem Antikörper, Antigen und zweitem Antikörper durch eine chemische Farbreaktion sichtbar gemacht, dagegen ist in der irt-PCR der zweite Antikörper über einen Streptavidin-Biotin-Komplex mit einer 246bp-langen doppelsträngigen DNA verbunden. Wenn der immunologische Komplex entsteht, kann diese Marker-DNA durch qPCR amplifiziert, detektiert und quantifiziert werden. Diese Methode ist etwa tausendmal sensibler als ein klassischer ELISA.
- Inverse PCR: Amplifikation unbekannter Genbereiche.
- Kolonie-PCR: Nachweis von bestimmten DNA-Sequenzen in Kolonien von Bakterien oder Pilzen; als DNA-Vorlage keine gereinigte Plasmid-DNA oder chromosomale DNA, sondern aus dem Kulturmedium entnommene Bakterienkolonien.
- Ligation-During-Amplification: Wird häufig zur Mutagenese von Plasmiden genutzt. Zirkuläre DNA kann amplifiziert werden, wodurch ein zusätzlicher Ligationsschritt entfällt.
- MassTag-PCR: Kombination einer PCR mit der Massenspektrometrie.
- Multiplex ligation-dependent probe amplification (MLPA): Variante der Multiplex-PCR (s. unten) zur gezielten Vermehrung mehrerer ähnlicher DNA-Sequenzen.
- Multiplex-PCR: Es werden mehr als ein Primerpaar für die Amplifikation eines bestimmten Gens[8] oder auch mehrerer Gene auf einmal verwendet. Die Multiplex-PCR spielt u. a. in der Diagnostik von Krankheiten eine Rolle, beispielsweise beim Lesch-Nyhan-Syndrom.[9] Ursache für das Lesch-Nyhan-Syndrom ist eine Mutation des HPRT1-Gens, das für eine Hypoxanthin-Guanin-Phosphoribosyltransferase codiert. Das Gen hat neun Exons, in Patienten mit dem Lesch-Nyhan-Syndrom kann eines dieser Exons fehlen. Durch eine Multiplex-PCR kann dies leicht entdeckt werden.
- Nested-PCR: Die nested (verschachtelte[10] bzw. geschachtelte) PCR eignet sich sehr gut, wenn nur sehr geringe Mengen der zu amplifizierenden DNA relativ zur Gesamtprobenmenge an DNA vorhanden sind. Hierbei werden zwei PCR hintereinander ausgeführt.[11] Durch die erste PCR wird – neben unerwünschten Sequenzbereichen infolge unspezifischer Bindung der Primer – der gewünschte Abschnitt der DNA (Amplikon) erzeugt. Letztere wird für eine zweite PCR als Matrize verwendet. Durch Primer, die an Bereichen innerhalb der ersten Matrize binden (downstream der ersten Primer), wird der gewünschte Sequenzbereich mit sehr hoher Spezifität generiert. Da auch die DNA-Region der Wahl zum zweiten Mal amplifiziert wurde, entsteht ausreichend DNA für weitere Prozedere. Anwendung findet die nested-PCR beispielsweise in der Gen-Diagnostik, in der Forensik (bei sehr wenig verwertbaren Spuren wie Haaren oder Bluttropfen wie im Kriminalfall JonBenét Ramsey) oder bei phylogenetischen Untersuchungen.[8] Auch Mikrochimärismus bei Leukozyten nach einer Bluttransfusion kann mittels nested-PCR nachgewiesen werden.
- PAN-PCR: Eine rechnerische Methode zur Gestaltung der Bakterientypisierung – Tests basierend auf Sequenzdaten des gesamten Genoms[12]
- Quantitative Echtzeit-PCR (qPCR oder real-time PCR): Wird benutzt, um die Menge des vervielfältigten DNA-Abschnitts zu bestimmen. Im Laborjargon wird fast nur der englische Begriff real-time PCR oder quantitative PCR verwendet, kurz qPCR oder missverständlich auch rt-PCR, was aber zu Verwechslungen mit dem länger etablierten Begriff RT-PCR mit vorgeschalteter reverser Transkription führt. Bei der real-time PCR wird der Reaktion ein zunächst inaktiver Fluoreszenzfarbstoff beigemischt, der durch die DNA-Produktion aktiv wird. (Zum Beispiel, weil er sich in die DNA einlagert (wie SYBR Green) oder weil ein Quencher, der die Fluoreszenz zunächst löscht, bei der Amplifikation entfernt wird.) Bei jedem Zyklus – also „in Echtzeit“ – wird die Fluoreszenz gemessen, woraus man auf die Menge der amplifizierten DNA schließen kann. Abhängig von der ursprünglichen Anzahl an Kopien wird ein gewisser Schwellenwert des Fluoreszenzsignals früher oder später (oder gar nicht) erreicht. Wegen dieser Zusatzinformation hat die real-time PCR den Beinamen „quantitative PCR (qPCR)“. Z. B. durch die Verwendung von Standardkurven erlaubt diese Technik auch eine absolute Quantifizierung (als Kopieanzahl pro Reaktion). Obwohl Geräte und farbstoffmarkierte Reagenzien teurer sind als bei der „klassischen“ Endpunkt-PCR, ist ein Sichtbarmachen der Amplifikate auf einem Gel nicht unbedingt nötig, so dass sich Arbeit und vor allem Zeit sparen lassen. Die Technik der qPCR lässt sich auch mit einer vorgeschalteten reversen Transkription (RT) kombinieren, z. B. um RNA-Viren nachzuweisen, dann spricht man von qRT-PCR.
- Reverse-Transkriptase-PCR (RT-PCR): Die Amplifikation von RNA (z. B. eines Transkriptoms oder eines RNA-Virus) erfolgt in einer Reverse-Transkriptions-PCR (engl. reverse transcription PCR) über eine reverse Transkription der RNA in DNA mit einer reversen Transkriptase. In Kombination mit einer Konzentrationsbestimmung in Echtzeit (qPCR) bezeichnet man die Reaktion als qRT-PCR.
- Touchdown-PCR: vermeidet eine Amplifizierung unspezifischer DNA-Sequenzen. In den ersten Synthese-Zyklen wird die Annealing-Temperatur nur knapp unterhalb der Denaturierungstemperatur gewählt. Damit ist die Primerbindung und somit auch das Amplifikat höchst spezifisch. In den weiteren Zyklen wird die Annealing-Temperatur herabgesetzt. Die Primer können jetzt zwar unspezifische Bindungen eingehen, allerdings verhindern die spezifischen Replikate der frühen Reaktion eine übermäßige Amplifikation der unspezifischen Sequenzen. Ein weiterer Vorteil ist eine enorme Steigerung der Amplifikatmenge. Diese abgewandelte PCR erlaubt somit eine starke und sehr spezifische DNA-Amplifikation.[13]
Anwendungsgebiete
[Bearbeiten | Quelltext bearbeiten]Die PCR kommt als Methode zur Detektion und Vervielfältigung von DNA-Abschnitten in einer Vielzahl von Anwendungsgebieten zum Einsatz.
Forschung
[Bearbeiten | Quelltext bearbeiten]- Das Klonieren eines Gens – nicht zu verwechseln mit dem Klonen eines ganzen Organismus – ist ein Vorgang, bei dem ein Gen aus einem Organismus isoliert und anschließend in einen anderen eingepflanzt wird. PCR wird oft benutzt, um das Gen zu vervielfältigen, das dann in einen Vektor (ein Vektor ist ein Mittel, mit dem ein Gen in einen Organismus verpflanzt werden kann), beispielsweise ein Plasmid (ein ringförmiges DNA-Molekül), eingefügt wird (siehe Abbildung). Die DNA kann anschließend in einen anderen Organismus eingesetzt werden, in dem das Gen oder sein Produkt besser untersucht werden kann. Das Exprimieren eines klonierten Gens kann auch zur massenhaften Herstellung nutzbarer Proteine wie z. B. Arzneimittel dienen.

Klonierung eines Gens mittels eines Plasmids:
(1) Chromosomale DNA von Organismus A.
(2) PCR.
(3) Mehrere Kopien eines einzelnen Gens von Organismus A.
(4) Einfügen des Gens in ein Plasmid.
(5) Plasmid mit dem Gen aus Organismus A.
(6) Einfügen des Plasmids in Organismus B.
(7) Vervielfältigung oder Expression des Gens, das aus Organismus A stammt, im Organismus B. - Mutagenese ist eine Möglichkeit, die Sequenz der Nukleotide (Basen) der DNA zu verändern. Es gibt Situationen, in denen man mutierte (veränderte) Kopien eines bestimmten DNA-Strangs benötigt, um die Funktion eines Gens zu bestimmen. Mutationen können in kopierte DNA-Sequenzen auf zwei grundsätzlich verschiedene Arten während des PCR-Prozesses eingefügt werden. Gezielte Mutagenese (engl. site-directed mutagenesis) erlaubt es dem Forscher, an spezifischen Stellen auf dem DNA-Strang Mutationen zu erzeugen. Meist wird dafür die gewünschte Mutation in die Primer integriert, die für die PCR verwendet werden. Bei der gezielten bzw. stellenspezifischen Mutagenese ist mindestens einer der Primer nicht hundertprozentig identisch mit der DNA, an die er sich anlagert. Während der Amplifikation wird so eine Mutation in das DNA-Fragment eingeführt. Zufällige Mutagenese (engl. random mutagenesis) beruht hingegen auf der Verwendung von fehlerträchtigen Polymerasen (bzw. Polymerasen ohne Mechanismus zur Fehlerkorrektur) während des PCR-Prozesses. Bei der zufälligen Mutagenese können Ort und Art der Mutationen nicht beeinflusst werden und müssen erst durch eine Sequenzierung identifiziert werden. Eine Anwendung der zufälligen oder gezielten Mutagenese ist die Analyse der Struktur-Funktions-Beziehungen eines Proteins. Nach der Veränderung der DNA-Sequenz kann man das entstandene Protein mit dem Original vergleichen und die Funktion aller Teile des Proteins bestimmen. Zudem können damit auch Funktionen der Nukleinsäure selbst (mRNA-Transport, mRNA-Lokalisation etc.) untersucht werden.
- Gängige Methoden der DNA-Sequenzierung (Bestimmung der Nukleotid-Abfolge von DNA) basieren auf Varianten der PCR. Die Illumina Sequenzierungsmethode (Sequenzierung mit Brückensynthese) beruht auf einer Festphasen-PCR, bei der die zu sequenzierende DNA zufällig fragmentiert, mit Oligonukleotiden (Adaptersequenzen) ligiert und über komplementäre Adaptersequenzen an einer Oberfläche fixiert wird. Bei der anschließenden PCR dienen die Adaptersequenzen als Primer. Zur Sequenzierung werden hierbei spezielle Nukleotide verwendet, die mit verschiedenfarbigen fluoreszierenden Markern versehen sind. Während der Amplifikation kann über die jeweils detektierte Farbe das eingebaute Nukleotid zugeordnet werden. Andere Sequenzierungsmethoden basieren auf der Emulsions-PCR. Beispiele sind die Zwei-Basen-Sequenzierung (engl. Sequencing by Oligo Ligation Detection, SOLiD) oder das Ionen-Halbleiter-DNA-Sequenzierungssystem (Ion Torrent Sequenzierungsmethode).[14]
Medizin und Diagnostik
[Bearbeiten | Quelltext bearbeiten]- Die Erkennung von Erbkrankheiten in einem vorliegenden Genom ist ein langwieriger und komplizierter Vorgang, der durch den Einsatz von PCR bedeutend verkürzt werden kann. Jedes Gen, das in Frage kommt, kann durch PCR mit den entsprechenden Primern amplifiziert (= vervielfältigt) und anschließend sequenziert werden (DNA sequenzieren heißt, die Sequenz der Nukleotide (oder Basen) der DNA zu bestimmen), um Mutationen aufzuspüren.
- Virale Erkrankungen können ebenfalls durch PCR erkannt werden, indem man die Virus-DNA vervielfältigt bzw. bei RNA-Viren diese RNA erst in DNA umschreibt und dann mittels PCR vervielfältigt (die RT-PCR).[15] Diese Analyse kann sofort nach der Infektion erfolgen, oft Tage oder Wochen vor dem Auftreten der Symptome. Erfolgt die Diagnose so früh, erleichtert das den Medizinern die Behandlung erheblich. Darüber hinaus wird die quantitative PCR auch für die Diagnostik verwendet, z. B. um die genaue Viruslast bei einer bekannten HIV-Infektion zu bestimmen, um die Entwicklung des Therapieerfolgs nachzuvollziehen.
- Die PCR kann auch zu Reihenuntersuchungen eingesetzt werden. So wird sie z. B. von Blutspendediensten zur Routineuntersuchung von Blutkonserven eingesetzt. Die Empfindlichkeit des PCR-Tests erlaubt es, Proben zu sogenannten Pools (z. B. 64 Einzelproben) zusammenzufassen. Ist der Test eines Pools positiv, wird die Anzahl der zusammengefassten Proben solange verringert (meistens halbiert), bis die verursachende Probe identifiziert ist.
- Zur sicheren Diagnostik und Absicherung von eventuell falsch-positiven Antigen-Schnelltests bei der COVID-19-Erkrankung wird die PCR ebenfalls eingesetzt.
Forensik, Paläontologie und Biologische Anthropologie
[Bearbeiten | Quelltext bearbeiten]- Der genetische Fingerabdruck ist ein DNA-Profil, das für jedes Individuum einzigartig ist. In der Forensik wird der genetische Fingerabdruck genutzt, um kleinste Spuren von an Tatorten gefundener DNA mit der DNA von Verdächtigen zu vergleichen. Als Proben können Blut, Sperma, Speichel, Hautzellen, oder Haare mit anhaftenden Zellen dienen. Die DNA wird aus den Zellkernen der in den Proben enthaltenen Körperzellen extrahiert, aufgereinigt und analysiert. Bei der Analyse wird die PCR genutzt, um spezielle DNA-Abschnitte zu vervielfältigen, die sich in nicht-codierenden Bereichen der DNA (junk DNA) befinden und aus Wiederholungen bestimmter kurzer Sequenzen (Tandemwiederholungen) bestehen. Die Anzahl der Tandemwiederholungen (und damit die Länge der Sequenzen) sind zwischen verschiedenen Individuen sehr variabel. Da mehrere unterschiedliche DNA-Abschnitte auf die jeweilige Anzahl der Tandemwiederholungen hin untersucht werden, entsteht ein individuelles Muster aus PCR-Produkten charakteristischer Längen.
- Abstammungsgutachten oder Vaterschaftstests basieren ebenfalls auf dem genetischen Fingerabdruck (siehe Abbildung).

Elektrophorese PCR- vervielfältigter DNA-Fragmente. (1) Vater. (2) Kind. (3) Mutter. Das Kind hat Teile der Fingerabdrücke der beiden Elternteile geerbt, wodurch es über einen eigenen, einzigartigen Fingerabdruck verfügt. - In der Molekularen Phylogenie zur Untersuchung evolutionärer Verwandtschaftsverhältnisse von Organismen.
- Analyse alter (fossiler) DNA: Da die PCR aus nur geringen DNA-Probemengen eine beliebige Menge von Material erzeugen kann, ist sie besonders für die sehr alte aDNA geeignet, die in der Natur nur noch in für Untersuchungen nicht mehr ausreichenden Mengen vorkommt. Dabei beruhen nahezu alle wissenschaftlichen Erkenntnisgewinne in Bezug auf die aDNA und somit viele seit langem ausgestorbener Arten auf der Methode der PCR.
Lebensmittelanalytik
[Bearbeiten | Quelltext bearbeiten]Die steigenden Anforderungen von Handel und behördlicher Lebensmittelüberwachung zur Aufklärung und Verhinderung von unlauterem Wettbewerb führten zum Einzug der Technologie in die Lebensmittelanalytik.[16] So kann die PCR zur Identifizierung von Gewürzen in komplexen Lebensmittelmatrizes herangezogen werden.[17] Sie kann auch zur Unterscheidung von Varietäten bei Edelkakao z. B. Criollo und Forastero eingesetzt werden.[18]
Literatur
[Bearbeiten | Quelltext bearbeiten]- C. R. Newton, A. Graham: PCR. Introduction to Scientific Techniques. 2. Auflage. ed. BIOS Scientific Publishers, Oxford 1997, ISBN 1-872748-82-1.
- R. K. Saiki, D. H. Gelfand, S. Stoffel, S. J. Scharf, R. Higuchi, G. T. Horn, K. B. Mullis, H. A. Erlich: Primer-Directed Enzymatic Amplification of DNA with a Thermostable DNA Polymerase (PDF; 1,2 MB). In: Science. 239.1988, S. 487–491, PMID 2448875, ISSN 0036-8075
- Kary B. Mullis, F. Faloona, S. Scharf, R. Saiki, G. Horn, H. Erlich: Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol. 1986;51 Pt 1:263-73.
- Patent US5656493A: System for automated performance of the polymerase chain reaction. Angemeldet am 18. Februar 1994, veröffentlicht am 12. August 1997, Anmelder: Perkin Elmer Corp, Erfinder: Kary B. Mullis et al.
- Kary B. Mullis: The polymerase chain reaction. Birkhäuser, Boston 1994, ISBN 3-7643-3607-2.
- Rabinow, Paul: Making PCR: A Story of Biotechnology, University of Chicago Press, 1996, ISBN 0-226-70146-8.
- D. Baltimore: RNA-dependent DNA polymerase in virions of RNA tumor viruses. In: Nature 226, 1970, S. 1209–1211.
Weblinks
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ The Nobel Prize in Chemistry 1993. Abgerufen am 24. November 2022 (amerikanisches Englisch).
- ↑ a b Patent US4683202A: Process for amplifying nucleic acid sequences. Angemeldet am 25. Oktober 1985, veröffentlicht am 28. Juli 1987, Anmelder: Cetus Corp, Erfinder: Kary B. Mullis.
- ↑ K. Kleppe et al.: Studies on polynucleotides. XCVI. Repair replications of short synthetic DNAs as catalyzed by DNA polymerases. In: J. Mol. Biol., 1971, Bd. 56, S. 341–361, PMID 4927950.
- ↑ Informationen der Nobelstiftung zur Preisverleihung 1993 an Kary B. Mullis (englisch).
- ↑ Eric S. Lander, Lauren M. Linton, Bruce Birren, Chad Nusbaum, Michael C. Zody: Initial sequencing and analysis of the human genome. In: Nature. Band 409, Nr. 6822, Februar 2001, ISSN 1476-4687, S. 860–921, doi:10.1038/35057062 (nature.com [abgerufen am 18. November 2020]).
- ↑ Debunking the 4 degree myth: PCR can be left at room temperature overnight – miniPCR. Abgerufen am 5. Juni 2018 (amerikanisches Englisch).
- ↑ Bernard J. Glick, Jack J. Pasternak, Cheryl L. Patten: Molecular Biotechnology: Principles and Applications of Recombinant DNA. John Wiley & Sons, 4. Auflage 2010, ISBN 978-1-55581-498-4, S. 359ff.
- ↑ a b Thomas M. Devlin: Textbook of Biochemistry with Clinical Correlations. John Wiley & Sons; 7. Auflage 2010, ISBN 978-0-470-28173-4, S. 269.
- ↑ JS. Chamberlain et al.: Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification. In: Nucleic Acids Research. 16(23), 1988, S. 11141–11156, PMID 3205741, PMC 339001 (freier Volltext)
- ↑ Cornel Mülhardt: Der Experimentator: Molekularbiologie / Genomics. 6. Auflage. Spektrum Akademischer Verlag, 2008, ISBN 978-3-8274-2036-7, S. 99.
- ↑ Ulrich Busch: Molekularbiologische Methoden in der Lebensmittelanalytik: Grundlegende Methoden und Anwendungen. Springer Verlag, Berlin 2010, ISBN 978-3-642-10715-3, S. 113.
- ↑ Journal of Clinical Microbiology Volume 51, Issue 3
- ↑ R Don, P Cox, B Wainwright, K Baker, J Mattick: Touchdown PCR to circumvent spurious priming during gene amplification. In: Nucleic Acids Res, 19(14);4008, PMID 1861999, PMC 328507 (freier Volltext)
- ↑ Hans-Joachim Müller, Daniel Ruben Prange: PCR - Polymerase-Kettenreaktion. Springer Berlin Heidelberg, Berlin, Heidelberg 2016, ISBN 978-3-662-48235-3, S. 140 ff., doi:10.1007/978-3-662-48236-0 (springer.com [abgerufen am 16. November 2020]).
- ↑ Carolina Scagnolari, Ombretta Turriziani, Katia Monteleone, Alessandra Pierangeli, Guido Antonelli: Consolidation of molecular testing in clinical virology. In: Expert Review of Anti-infective Therapy. Band 15, Nr. 4, 3. April 2017, ISSN 1478-7210, S. 387–400, doi:10.1080/14787210.2017.1271711 (tandfonline.com [abgerufen am 25. September 2020]).
- ↑ M. Fischer and I. Haase: PCR in der Lebensmittelanalytik, GIT Labor-Fachzeitschrift 3/06 (2006).
- ↑ F. Focke, I. Haase and M. Fischer: Trends in der Lebensmittelanalytik: DNA-basierte Wege zur Identifizierung von Gewürzen, Nachrichten aus der Chemie, 57, 1017–1020 (2009).
- ↑ I. Haase, M. Fischer: Differenzierung von Theobroma cacao und Theobroma grandiflorum mittels PCR. In: Journal für Verbraucherschutz und Lebensmittelsicherheit. Band 2, Nr. 4, 1. November 2007, ISSN 1661-5751, S. 422–428, doi:10.1007/s00003-007-0252-1.

