Proliferating-Cell-Nuclear-Antigen
| Proliferating-Cell-Nuclear-Antigen | ||
|---|---|---|
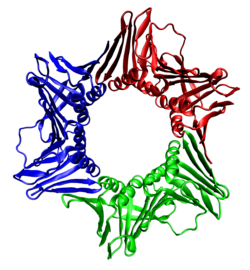
| ||
| Bändermodell des PCNA-Trimer nach PDB 1AXC | ||
|
Vorhandene Strukturdaten: s. UniProt | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 261 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotrimer | |
| Bezeichner | ||
| Gen-Name | PCNA | |
| Externe IDs | ||
| Vorkommen | ||
| Homologie-Familie | Hovergen | |
| Übergeordnetes Taxon | Eukaryoten[1] | |
| Übergeordnet |
|---|
| DNA-Replikationsfaktor-C-Komplex |
| Gene Ontology |
| QuickGO |
Proliferating-Cell-Nuclear-Antigen (PCNA) ist ein Protein, das während der eukaryotischen DNA-Replikation die DNA als Ring umgibt (so genanntes Ringklemmenprotein). Nur durch PCNA ist es möglich, dass während der S-Phase des Zellzyklus die gesamte DNA mit hoher Geschwindigkeit und ohne größere Unterbrechungen vervielfältigt werden kann.
Genetik
[Bearbeiten | Quelltext bearbeiten]Das PCNA-Gen liegt beim Menschen auf Chromosom 20. Es sind bislang zwei Transkriptionsvarianten bekannt, die das gleiche Protein kodieren.[2] Der Promotorbereich enthält Bindungsstellen für den Transkriptionsfaktor E2F. Pseudogene dieses Gens wurden auf Chromosom 4 und dem X-Chromosom entdeckt.[3]
Struktur
[Bearbeiten | Quelltext bearbeiten]PCNA besteht aus 261 Aminosäuren und besitzt ein molekulares Gewicht von ca. 28,7 kDa. Das Protein besteht aus drei identischen Untereinheiten (Homotrimer), die einen Ring bilden und durch insgesamt 12 symmetrisch gelegene α-Helices an doppelsträngige DNA (ds-DNA) binden können.[2][4]
Funktion
[Bearbeiten | Quelltext bearbeiten]Im Rahmen der eukaryotischen DNA-Replikation werden zunächst mit Hilfe der Primase (RNA-Polymerase) kurze RNA-Primer auf den entwundenen Matrizenstrang synthetisiert (De-novo-Synthese). Das dabei gebildete Primer-Matrize-Hybrid wird direkt an die assoziierte DNA-Polymerase α weitergereicht, welche den RNA-Primer am 3’-Ende verlängert und die DNA-Synthese startet. Die Primer bestehen somit am 5’-Ende aus 8–10 Ribonucleotiden (RNA) und am 3’-Ende aus ca. 20 Deoxynucleotiden (DNA). Das 3’-Ende des Primers bildet somit einen Abschnitt mit ds-DNA. Dieser Übergang von der ds-DNA am Primer-Ende zur einzelsträngigen DNA (ss-DNA) des Matrizenstrangs wird vom Replication Factor C (RF-C) erkannt, welcher die PCNA-Ringklemme aufsetzt. Die replikativen DNA-Polymerasen δ (am Folgestrang) und ε (am Leitstrang) binden an PCNA und verlängern den Primer – entweder bis sie auf das nächste Okazaki-Fragment treffen (Folgestrang) oder bis zur nächsten Replikationsblase (Leitstrang).[5] Während der gesamten Elongationsphase bleibt PCNA an die DNA-Polymerase gebunden und verhindert deren Dissoziation vom DNA-Strang. Erst wenn das letzte Desoxynucleotid eingefügt ist, fällt die Polymerase ab und kann anderweitig wieder an PCNA binden.[6]
Im Falle von DNA-Schäden bewirkt der Tumorsuppressor p53 die verstärkte Bildung des CDK-Inhibitors p21. p21 kann unter anderem an die PCNA-Ringklemme binden, was zum sofortigen Stopp der Replikation führt. Dies gibt geschädigten Zellen Zeit für die DNA-Reparatur, wodurch verhindert wird, dass verändertes Erbgut an Tochterzellen weitergegeben wird.[6]
PCNA bindet auch an die DNA-Polymerase δ, wenn diese für die DNA-Reparatur rekrutiert wird. Dies geschieht vor allem, um die Einzelstranglücken, die im Rahmen der Nukleotidexzisionsreparatur (NER) entstehen, zu füllen.[6] In diesem Fall ist PCNA an Lysin 164 monosumoyliert, wohingegen es während der Replikation einen Ubiquitin-Baustein an der gleichen Stelle trägt.[7]
Der Ladungsfaktor RF-C gehört zu den ATPasen der Klasse AAA+.[6]
Evolution
[Bearbeiten | Quelltext bearbeiten]PCNA ist funktionell analog zur beta-clamp bei Bakterien. Die beta-clamp bindet ebenfalls mit 12 symmetrisch gelegenen α-Helices an die DNA, ist jedoch – anders als PCNA – ein Homodimer. Dieses Homodimer besteht aus zwei Molekülen der β-Untereinheit von DNA-Polymerase III. Die beta-clamp wird vom sogenannten γ-Komplex auf die DNA geladen. Der γ-Komplex mit seinen fünf Untereinheiten kann als Analogon zu RF-C bei den Eukaryoten angesehen werden.[6]
Weblinks
[Bearbeiten | Quelltext bearbeiten]- reactome: Loading of PCNA - Sliding Clamp Formation
- reactome: Interaction between FEN1 and PCNA
- Blackburn/Seidel/reactome: Formation of Processive Complex on the C-strand of the telomere
- Blackburn/Seidel/reactome: Disassociation of Processive Complex and Completed Telomere End
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Orthologe bei OMA
- ↑ a b GeneCards: PCNA
- ↑ Egelkrout EM, Mariconti L, Settlage SB, Cella R, Robertson D, Hanley-Bowdoin L (2002). "Two E2F elements regulate the proliferating cell nuclear antigen promoter differently during leaf development". Plant Cell 14 (12): 3225–36. doi:10.1105/tpc.006403. PMID 12468739.
- ↑ H. Lodish et al.: Molecular Cell Biology, sixth Edition, W.H. Freeman and Company, New York 2008
- ↑ Joachim Rassow, Karin Hauser, Roland Netzker, Rainer Deutzmann: "Duale Reihe: Biochemie" S. 428, 3. vollständig überarbeitete und erweiterte Auflage, Thieme Verlag 2012, ISBN 978-3-13-125353-8
- ↑ a b c d e Rolf Knippers: Molekulare Genetik, 9. komplett überarbeitete Auflage, Thieme Verlag 2006, ISBN 3-13-477009-1
- ↑ Hoege C, Pfander B, Moldovan GL, Pyrowolakis G, Jentsch S: RAD6-dependent DNA repair is linked to modification of PCNA by ubiquitin and SUMO. In: Nature. 419. Jahrgang, Nr. 6903, September 2002, S. 135–41, doi:10.1038/nature00991, PMID 12226657.