Transcription-Translation Feedback Loop
Der Transcription-Translation Feedback Loop (TTFL), zu deutsch Transkriptions-Translations-Rückkopplungsschleife, ist ein zelluläres Modell zur Erklärung circadianer Rhythmen in Verhalten und Physiologie. Der TTFL ist in vielen Arten konserviert und reguliert sich automatisch. Dabei wird die Transkription von bestimmten Genen, den sogenannten Clock-Genen („Uhr“-Genen), durch ihre eigenen Proteinprodukte reguliert.
Entdeckung
[Bearbeiten | Quelltext bearbeiten]Circadiane Rhythmen sind bereits seit Jahrhunderten bekannt. So bemerkte der französische Astronom Jean Jacques d’Ortous de Mairan bereits 1729 die periodische Bewegung von Pflanzenblättern der Mimosen innerhalb von 24 Stunden. Die Wissenschaft hat jedoch erst vor kurzem begonnen, die zellulären Mechanismen aufzudecken, die für den Antrieb der beobachteten circadianen Rhythmen verantwortlich sind. Die zelluläre Basis von circadianen Rhythmen wird durch die Tatsache gestützt, dass Rhythmen in einzelligen Organismen beobachtet wurden.[1]
Ab den 1970er Jahren zeigten Experimente von Ron Konopka et al., bei denen Methoden der Vorwärts-Genetik (englisch: forward genetics) zur Induzierung von Mutationen eingesetzt wurden, dass Arten von Drosophila melanogaster mit veränderten Genen, den sogenannten period-Genen (per), ebenfalls eine veränderte Periodizität aufwiesen. Mit der Verbesserung der genetischen und molekularbiologischen experimentellen Instrumente identifizierten die Forscher weitere Gene, die für die Aufrechterhaltung eines normalen rhythmischen Verhaltens verantwortlich sind. So entstand das Konzept, dass interne Rhythmen durch eine kleine Untergruppe von core clock-Genen modifiziert werden. Paul Hardin et al. schlugen im Jahr 1990 als erste Gruppe vor, dass der Mechanismus, der diese Rhythmen antreibt, eine negative Rückkopplungsschleife ist.
Nachfolgende wichtige Entdeckungen bestätigten dieses Modell. Insbesondere die Experimente unter der Leitung von Thomas K. Darlington und Nicholas Gekakis Ende der 1990er-Jahre identifizierten CLOCK-Proteine und charakterisierten ihre Methoden in Drosophila bzw. Mäusen. Aus diesen Experimenten entstand das TTFL-Modell, das mittlerweile zum vorherrschenden Paradigma für die Erklärung des circadianen Verhaltens für eine Vielzahl von Arten geworden ist.[2]
Allgemeine Mechanismen der TTFL
[Bearbeiten | Quelltext bearbeiten]Die TTFL ist eine negative Rückkopplungsschleife, in der Clock-Gene durch ihre Proteinprodukte reguliert werden. Im Allgemeinen umfasst die TTFL zwei Rückkopplungsschleifen: positive regulatorische Elemente, die die Transkription fördern und Proteinprodukte, die die Transkription unterdrücken. Wenn ein positives regulatorisches Element an einen Clock-Promotor bindet, schreitet die Transkription voran, was zur Bildung eines mRNA-Transkripts führt. Anschließend erfolgt die Translation, was zu einem Proteinprodukt führt. Es gibt charakteristische Verzögerungen zwischen mRNA-Transkriptakkumulation, Proteinakkumulation und Gen-Silencing aufgrund der Translationsdynamik, posttranslationalen Modifikation, Proteindimerisierung und intrazellulärem Transport zum Zellkern.[3]
Artübergreifende Proteine, die sich am TTFL beteiligen, enthalten gemeinsame Strukturmotive wie PAS-Domänen, die an Protein-Protein-Wechselwirkungen beteiligt sind und bHLH-Domänen, die an der DNA-Bindung beteiligt sind.[4]
Modelle
[Bearbeiten | Quelltext bearbeiten]Das Vorhandensein der TTFL ist bei allen Tierarten stark konserviert. Viele der am Prozess beteiligten Akteure haben sich jedoch im Laufe der Entwicklungszeit innerhalb verschiedener Arten verändert. Beim Vergleich von Pflanzen, Tieren, Pilzen und anderen Eukaryoten gibt es Unterschiede in den Genen und Proteinen, die an der TTFL beteiligt sind. Dies deutet darauf hin, dass sich eine „Uhr“, die dem TTFL-Modell folgt, während der Existenz des Lebens mehrmals entwickelt hat.[5]
| Drosophila melanogaster | |
|---|---|
| Positive Regulatoren | CYC, Clock |
| Negative Regulatoren | TIM, PER |
| Säugetiere | |
| Positive Regulatoren | BMAL1, CLOCK |
| Negative Regulatoren | PER1, PER2, CRY1, CRY2 |
| Neurospora | |
| Positive Regulatoren | WC-1, WC-2 |
| Negative Regulatoren | FRQ |
Drosophila melanogaster
[Bearbeiten | Quelltext bearbeiten]Der TTFL wurde erstmals in Drosophila entdeckt und das System weist mehrere Gemeinsamkeiten mit der TTFL von Säugetieren auf. Die Transkription der Clock-Gene period (per) und timeless (tim) wird initiiert, wenn die positiven Proteinelemente CYCLE (dCYC) und CLOCK (dCLK) ein Heterodimer bilden und E-Box-Promotoren binden, wodurch die Transkription initiiert wird. Während des Tages wird das Proteinprodukt TIM abgebaut; Belichtung erleichtert das Binden des Proteins CRY an TIM, wodurch TIM ubiquitiniert und schließlich abgebaut wird.[6] In der Nacht können TIM und PER Heterodimere bilden und sich langsam im Cytoplasma ansammeln, wo PER durch die Kinase DBT (double-time protein) phosphoryliert wird. Die posttranskriptionelle Modifikation mehrerer Phosphatgruppen dient zum Abbau des Komplexes und zur Erleichterung der Lokalisation des Komplexes im Zellkern. Im Zellkern bindet das PER-TIM-Dimer an das CYC-CLK-Dimer, wodurch das CYC-CLK-Dimer aus den E-Boxen freigesetzt und die Transkription inhibiert wird. Sobald sich PER und TIM zersetzen, können CYC-CLK-Dimere die E-Boxen erneut binden, um die Transkription zu initiieren und die negative Rückkopplungsschleife zu schließen.[7]
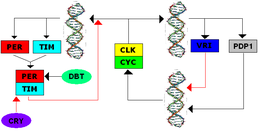
Sekundäre Rückkopplungsschleifen interagieren mit der primären Rückkopplungsschleife. CWO (Clockwork Orange Drosophila protein) bindet die E-Boxen als direkten Konkurrenten von CYC-CLK und hemmt so die Transkription. PDP1ε (par domain protein 1ε) ist ein Rückkopplungsaktivator und VRI (vrille) ist ein Rückkopplungsinhibitor des Clock-Promotors; ihre Expression wird durch dCLK-dCYC aktiviert. Das Ecdyson-induced Protein 75 (E75) hemmt die Clock-Expression und aktiviert sich zeitabhängig pro Transkription. Sämtliche sekundäre Schleifen verstärken den primären TTFL.[7]
Cryptochrom in Drosophila ist ein Blaulicht-Photorezeptor, der den Abbau von TIM auslöst und indirekt dazu führt, dass die Taktphase zurückgesetzt und die Expression des per-Gens erneut gefördert wird.[7]
Säugetiere
[Bearbeiten | Quelltext bearbeiten]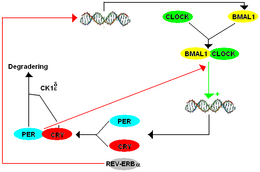
Das TTFL-Modell der Säugetiere enthält viele Komponenten, die Homologen derjenigen sind, die in Drosophila gefunden wurden. Der TTFL der Säugetiere arbeitet so, dass BMAL1 mit CLOCK ein Heterodimer bildet, um die Transkription von mPer und cryptochrom (cry) zu initiieren. Es gibt drei Paraloge oder historisch ähnliche Gene, die heutzutage als Duplikation des period-Gens in Säugetieren auftreten, die als mPer1, mPer2 und mPer3 aufgeführt sind. Es gibt auch zwei Paraloge von Cryptochrom bei Säugetieren. PER- und CRY-Proteine bilden ein Heterodimer und die Phosphorylierung von PER durch CK1δ (Casein kinase I isoform delta) und CK1ε (Casein kinase 1 isoform epsilon) reguliert die Lokalisierung des Dimers zum Zellkern. Im Zellkern reguliert PER-CRY die Transkription ihrer verwandten Gene negativ, indem es BMAL1-CLOCK bindet und sie vom E-Box-Promotor freisetzt.[7]
Obwohl die mPer-Paraloge als funktionale Orthologen von dPer zusammenarbeiten, haben sie jeweils eine besondere Funktion. mPer1 und mPer2 sind für die „Uhrfunktion“ im Gehirn notwendig, während mPer3 nur im circadianen Rhythmus peripherer Gewebe eine erkennbare Rolle spielt. Beim Gen-Knockout von mPer1 oder mPer2 bewirkt eine Änderung der Freilauf-Periode (τ), wobei mPer1 mit einer kürzeren Periode und mPer2 mit einer längeren Periode im Vergleich zur ursprünglichen Periode ausfällt, bevor es schließlich zu einer Arrhythmie kommt. In ähnlicher Weise führen mCry1-Knockouts zu einer verkürzten Periode und mCry2-Knockouts zu einer verlängerten Periode, wobei ein doppelter mCry1- oder mCry2-Knockout zu Arrhythmie führt.[7]
Es gibt auch sekundäre Schleifen bei Säugetieren, obwohl diese komplexer sind als bei Drosophila. Wie bei CWO in Drosophila unterdrücken Dec1 (Deleted in esophageal cancer 1) und Dec2 (Deleted in esophageal cancer 2) die mPer-Expression durch Binden von E-Boxen, wodurch der Proteinkomplex CLOCK-BMAL1 daran gehindert wird, sich an die E-Boxen zu binden. Die Rezeptoren Rev-erb und ROR (RAR-related orphan receptor) spielen eine ähnliche Rolle wie PDP1ε und VRI in Drosophila, außer dass sie den CLOCK-Bindungspartner BMAL1 regulieren, anstatt CLOCK direkt zu regulieren. DBP (D site of albumin promoter (albumin D-box) binding protein) und E4BP4 (E4 Promoter-Binding Protein 4) binden an die D-Box-Promotorsequenz, um die mPer-Expression zu regulieren.[7]
Die Art und Weise, wie diese Gene mit Drosophila melanogaster in Beziehung stehen, wird in der Funktion jedes einzelnen Gens gesehen und in der Art und Weise, in der sie sich evolutionär verändert haben. BMAL1 ist ein Ortholog von CYCLE. Dies bedeutet, dass BMAL1 und CYCLE scheinbar eine gemeinsame Vorgeschichte haben, jedoch bei verschiedenen Arten vorkommen. Ein weiteres Beispiel für die Parallelen zwischen Drosophila melanogaster und Säugetieren findet sich auch in cry und mPer, da es sich um funktionelle Orthologe von per und tim handelt.[7]
Neurospora
[Bearbeiten | Quelltext bearbeiten]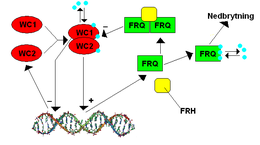
Das Gen frequency (frq) in Neurospora wurde 1979 von J. F. Feldman und seinen Kollegen als das damals zweite bekannte Clock-Gen identifiziert. FRQ wurde erstmals 1989 von C. R. McClung und seinen Kollegen geklont. Dieses Gen war von besonderem Interesse, da seine Expression im Vergleich zu anderen bekannten mikrobiellen Genen sehr komplex ist.
Zwei positive Regulatorproteine, Wc-1 (white collar 1 protein) und Wc-2 (white collar 2 protein), binden den frq-Promotor, der als Clock Box bezeichnet wird, während der späten subjektiven Nacht, um die Transkription zu aktivieren. Licht ist auch wichtig, um die FRQ-Expression zu induzieren. WC-1 ist ein Photopigment und Licht ermöglicht WC-1 und WC-2, dass sie sich an einen anderen Promotor binden können, der als proximales Lichtreaktionselement (PLRE) bezeichnet wird. Das FRQ-Protein reguliert die Aktivität von WC-1 und WC-2 negativ. Mehrere Kinasen (CK1-, CK2- und Checkpoint kinase 2-like) und Phosphatasen (PP1 und PP2A) regulieren die Fähigkeit von FRQ, sich in den Zellkern zu verlagern, sowie die Stabilität von FRQ, WC-1 und WC-2.[8]
Arabidopsis
[Bearbeiten | Quelltext bearbeiten]Das erste TTFL-Modell wurde für Arabidopsis im Jahr 2001 vorgeschlagen und umfasste drei MYB-Transkriptionsfaktoren, LHY (Late Elongated Hypocotyl), CCA1 (Circadian Clock Associated 1) und TOC1 (Timing of CAB expression 1). CCA1 und LHY werden am Morgen exprimiert und interagieren miteinander, um die Expression von TOC1 zu unterdrücken. Die CCA1- und LHY-Expression nimmt in der Dunkelheit ab, sodass TOC1 exprimiert und die CCA1- und LHY-Expression negativ reguliert wird. CCA1 und LHY können sich auch an ihren eigenen Promotor binden, um ihre eigene Transkription zu unterdrücken.[9]
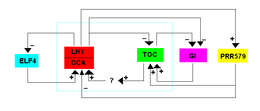
Eine zweite Schleife besteht aus PRR9, PRR7 und PRR5, die alle Homologe von TOC1 sind und die CCA1- und LHY-Expression unterdrücken. Diese PRR-Gene werden direkt von LHY und TOC1 unterdrückt. Diese Gene werden auch durch den Evening Complex (EC) reguliert, der aus LUX ARRHYTHMO (LUX), EARLY FLOWERING 3 (ELF3) und EARLY FLOWERING 4 (ELF4) besteht. LUX ist ein Transkriptionsfaktor mit einer ähnlichen Funktion wie MYB, während ELF3 und ELF4 Kernproteine sind, deren Funktionen unbekannt sind. Der Evening complex fördert indirekt die Expression von LHY und CCA1, wodurch die Transkription der eigenen Komponenten unterdrückt wird. Da dieses Modell aus zwei Hemmungen besteht, die zu einer Aktivierung führen, wird es auch als Repressilator bezeichnet.[9]
Eine kürzlich entdeckte Schleife umfasst die reveille Genfamilie, die morgens exprimiert wird und die Transkription von „Abend-Genen“ wie PRR5, TOC1, LUX und ELF4 induziert. Sobald die resultierenden Proteine translatiert sind, unterdrücken PRR9, PRR7 und PRR5 das Gen RVE8. RVE8 interagiert auch mit den „Morgen-Komponenten“ LNK1, 2, 3 und 4 (LNK: night light-inducible and clock-regulated), wobei LNKs das Gen RVE8 entweder antagonisieren oder co-aktivieren.[9]
Obwohl das Protein GIGANTEA (GI) nicht als Kernbestandteil des Arabidopsis-TFL-Modells bekannt ist, wird es von CCA1, LHY und TOC1 unterdrückt. Zusätzlich aktiviert GI die CCA1- und LHY-Expression.[9]
Cyanobakterien
[Bearbeiten | Quelltext bearbeiten]Studien zur „Cyanobakterien-Uhr“ führten zur Entdeckung von drei wesentlichen Clock-Genen: KaiA, KaiB und KaiC. Anfänglich wurde angenommen, dass diese Proteine dem TTFL-Modell ähneln, das für Eukaryoten vorgeschlagen wurde, da es ein tägliches Muster in Bezug auf mRNA- und Proteinhäufigkeit und Phosphorylierungsgrad gibt, sowie eine negative Rückkopplung von Proteinen auf ihre verwandten Gene, ein Zurücksetzen der Taktphase als Reaktion auf eine KaiC-Überexpression und eine modifizierte Kai-Aktivität durch Wechselwirkungen gab.[10]
Jedes dieser Ergebnisse stimmte mit dem damaligen Verständnis des TTFL überein. Spätere Studien kamen jedoch zu dem Schluss, dass posttranslationale Modifikationen wie die Phosphorylierung für die Taktsteuerung wichtiger sind. Wenn Promotoren für die Kai-Proteine durch unspezifische Promotoren ersetzt wurden, trat keine Unterbrechung der zentralen Rückkopplungsschleife auf, wie dies zu erwarten war, wenn eine Hemmung durch die Rückkopplung der Proteine auf ihre spezifischen Promotoren auftrat. Infolgedessen wurde festgestellt, dass das TTFL-Modell für Cyanobakterien weitgehend ungenau ist. Die Transkriptionsregulation ist nicht der zentrale Prozess, der den Rhythmus von Cyanobakterien antreibt. Obwohl Transkriptions- und Translationsregulierung vorhanden sind, wurden sie eher als Auswirkungen der „Uhr“ als für die Uhrfunktion notwendig erachtet.[11]
Alternativen zum TTFL-Modell
[Bearbeiten | Quelltext bearbeiten]Es wurden auch posttranslationale Rückkopplungsschleifen (PTFL) aufgedeckt, die an der Regulierung von Clock-Genen beteiligt sind und häufig in Verbindung mit dem TTFL-Modell arbeiten. Sowohl bei Säugetieren als auch bei Pflanzen regulieren posttranslationale Modifikationen wie Phosphorylierung und Acetylierung die Häufigkeit und/oder die Aktivität von Clock-Genen und -Proteinen. Beispielsweise wurde gezeigt, dass die Phosphorylierungsgrade von TTFL-Komponenten rhythmisch variieren. Diese posttranslationalen Modifikationen können als Degradationssignale, Bindungsregulatoren und Signale für die Rekrutierung zusätzlicher Transkriptionsfaktoren dienen.[12]
Insbesondere zeigen Cyanobakterien rhythmische Veränderungen der Phosphorylierung nach 24 Stunden in einer von Transkription und Translation unabhängigen Rückkopplungsschleife: circadiane Rhythmen der Phosphorylierung werden beobachtet, wenn die Kai-Proteine der Rückkopplungsschleife mit ATP in ein Reagenzglas gegeben werden, unabhängig von anderen zellulären Mechanismen. Es ist allgemein anerkannt, dass dieses posttranslationale System aus drei Proteinen der Hauptoszillator ist, der notwendig und ausreichend ist, um den täglichen Rhythmus voranzutreiben.[13]
Zusätzlich zum Kai-System in Cyanobakterien wurde gezeigt, dass die Oxidation von Peroxiredoxin unabhängig von der Transkription und Translation sowohl in roten Säugetierblutzellen als auch in Zellen der Grünalge Ostreococcus tauri auftritt. Es wurde festgestellt, dass dieses System in vielen Organismen konserviert ist.[14] Es ist nicht klar, ob das Peroxiredoxin-System mit TTFL-basierten Uhren interagiert oder selbst Teil einer neuen PTFL-basierten Uhr ist. Beide Befunde implizieren jedoch, dass in einigen Organismen oder Zelltypen PTFL ausreichen, um den circadianen Rhythmus zu steuern.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ D. Mergenhagen: Circadian rhythms in unicellular organisms. In: Current topics in microbiology and immunology. Band 90, 1980, S. 123–147, doi:10.1007/978-3-642-67717-5_6, PMID 6775877 (Review).
- ↑ Lisa Wulund, Akhilesh B. Reddy: A brief history of circadian time: The emergence of redox oscillations as a novel component of biological rhythms. In: Perspectives in Science. 6, 2015, S. 27, doi:10.1016/j.pisc.2015.08.002.
- ↑ M. H. Hastings, E. S. Maywood, J. S. O’Neill: Cellular circadian pacemaking and the role of cytosolic rhythms. In: Current Biology. Band 18, Nummer 17, September 2008, S. R805–R815, doi:10.1016/j.cub.2008.07.021, PMID 18786386 (Review).
- ↑ J. C. Dunlap, J. J. Loros, Y. Liu, S. K. Crosthwaite: Eukaryotic circadian systems: cycles in common. In: Genes to cells : devoted to molecular & cellular mechanisms. Band 4, Nummer 1, Januar 1999, S. 1–10, PMID 10231388 (Review).
- ↑ A. S. Loudon: Circadian biology: a 2.5 billion year old clock. In: Current Biology. Band 22, Nummer 14, Juli 2012, S. R570–R571, doi:10.1016/j.cub.2012.06.023, PMID 22835791.
- ↑ T. Yoshii, C. Hermann-Luibl, C. Helfrich-Förster: Circadian light-input pathways in Drosophila. In: Communicative & integrative biology. Band 9, Nummer 1, 2016 Jan-Feb, S. e1102805, doi:10.1080/19420889.2015.1102805, PMID 27066180, PMC 4802797 (freier Volltext) (Review).
- ↑ a b c d e f g T. S. Andreani, T. Q. Itoh, E. Yildirim, D. S. Hwangbo, R. Allada: Genetics of Circadian Rhythms. In: Sleep medicine clinics. Band 10, Nummer 4, Dezember 2015, S. 413–421, doi:10.1016/j.jsmc.2015.08.007, PMID 26568119, PMC 4758938 (freier Volltext) (Review).
- ↑ J. C. Dunlap, J. J. Loros, H. V. Colot, A. Mehra, W. J. Belden, M. Shi, C. I. Hong, L. F. Larrondo, C. L. Baker, C. H. Chen, C. Schwerdtfeger, P. D. Collopy, J. J. Gamsby, R. Lambreghts: A circadian clock in Neurospora: how genes and proteins cooperate to produce a sustained, entrainable, and compensated biological oscillator with a period of about a day. In: Cold Spring Harbor symposia on quantitative biology. Band 72, 2007, S. 57–68, doi:10.1101/sqb.2007.72.072, PMID 18522516, PMC 3683860 (freier Volltext) (Review).
- ↑ a b c d S. E. Sanchez, S. A. Kay: The Plant Circadian Clock: From a Simple Timekeeper to a Complex Developmental Manager. In: Cold Spring Harbor perspectives in biology. Band 8, Nummer 12, Dezember 2016, S. , doi:10.1101/cshperspect.a027748, PMID 27663772, PMC 5131769 (freier Volltext) (Review).
- ↑ C. H. Johnson, T. Mori, Y. Xu: A cyanobacterial circadian clockwork. In: Current Biology. Band 18, Nummer 17, September 2008, S. R816–R825, doi:10.1016/j.cub.2008.07.012, PMID 18786387, PMC 2585598 (freier Volltext) (Review).
- ↑ Ben Sheredos: Scientific Diagrams as Traces of Group-Dependent Cognition: A Brief Cognitive-Historical Analysis. In: Cognitive Science Society. eScholarship, 2013, abgerufen am 28. Oktober 2019.
- ↑ S. Kojima, D. L. Shingle, C. B. Green: Post-transcriptional control of circadian rhythms. In: Journal of cell science. Band 124, Pt 3Februar 2011, S. 311–320, doi:10.1242/jcs.065771, PMID 21242310, PMC 3021995 (freier Volltext) (Review).
- ↑ J. M. Hurley, J. J. Loros, J. C. Dunlap: Circadian Oscillators: Around the Transcription-Translation Feedback Loop and on to Output. In: Trends in Biochemical Sciences. Band 41, Nummer 10, 10 2016, S. 834–846, doi:10.1016/j.tibs.2016.07.009, PMID 27498225, PMC 5045794 (freier Volltext) (Review).
- ↑ S. A. Brown, E. Kowalska, R. Dallmann: (Re)inventing the circadian feedback loop. In: Developmental cell. Band 22, Nummer 3, März 2012, S. 477–487, doi:10.1016/j.devcel.2012.02.007, PMID 22421040 (Review).