Rhodopseudomonas
| Rhodopseudomonas | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Rhodopseudomonas | ||||||||||||
| Czurda and Maresch 1937 |
Rhodopseudomonas ist eine Gattung von Bakterien. Sie zählt zu den Purpurbakterien und führt eine anoxygene Photosynthese aus. Anoxygen bedeutet hier, dass im Gegensatz zur oxygenen Photosynthese von Cyanobakterien und Pflanzen kein Sauerstoff entsteht.
Erscheinungsbild
[Bearbeiten | Quelltext bearbeiten]Die Zellen von Rhodopseudomonas sind stäbchenförmig und besitzen Flagellen. Die Flagellen liegen entweder an einem Zellende (polar) oder auch mehr in der Nähe eines Zellendes (subpolar). Es erfolgt polares Wachstum, Knospenbildung und asymmetrische Zellteilung. Die Knospenbildung erfolgt ohne Hyphen. In älteren Kulturen werden auch rosettenartigen Zellaggregate gebildet.[1] So kann man bei Rhodopseudomonas palustris ein asymmetrisch geformtes Stäbchen mit einer dunkel erscheinenden Anhäufung von Thylakoiden an einem Pol sehen. Der gegenüberliegende Pol ist heller gefärbt, hier befindet sich die Wachstumszone.[2]
Sporen werden nicht gebildet. Der Gram-Test fällt negativ aus.
Wachstum und Stoffwechsel
[Bearbeiten | Quelltext bearbeiten]Rhodopseudomonas ist fakultativ anaerob und zählt zu den Nicht-Schwefelpurpurbakterien. Zur Energiegewinnung können die Arten die Photosynthese durchführen. Die Arten sind hierbei in der Regel photoorganotroph, sie benötigen zusätzliche organische Verbindungen als Elektronendonatoren für die Photosynthese. R. palustris u. R. julia sind auch in der Lage, vollkommen photoautotroph zu wachsen, d. h. sie benötigen keine weitere organischen Verbindungen für den Energiestoffwechsel.
Bei der Photosynthese von Rhodopseudomonas kann z. B. Thiosulfat als Elektronendonator ("Elektronenspender") dienen, bei der Photosynthese von Pflanzen und Grünalgen ist der Elektronendonator Wasser (H2O).
Wenn Rhodopseudomonas palustris im Licht kultiviert wird, bilden sich innere Membranstrukturen. Hier findet die Photosynthese statt. Die Membranen liegen parallel zur Zytoplasmamembran. Hier befinden sich die Bacteriochlorophylle und verschiedene Carotinoide, sowie Ubichinon-10, Cytochrome und weitere am Elektronentransport beteiligte Verbindungen.[2] Bei Rhodopseudomonas ist unter Ausschluss von Licht ebenfalls Wachstum möglich (sogenanntes chemotrophes Wachstum). Dies kann unter geringen Sauerstoffgehalt (mikrooxisch) und oxischen Bedingungen („normaler Sauerstoffgehalt“) erfolgen.[1]
Einige Arten von Rhodopseudomonas können freien, molekularen Stickstoff (N2) fixieren (s.h. Stickstofffixierung). Die Art Rhodopseudomonas rhenobacensis ist in der Lage, Nitrat zu Nitrit zu reduzieren. Im Labor häuft sich das gebildete Nitrit dann in dem Medium an.[1]
Einige Arten sind auch in der Lage, zur Energiegewinnung Schwefelwasserstoff (H2S) oder Schwefel zu Sulfat zu oxidieren. Hierzu zählt z. B. Rhodopseudomonas julia. Das Zwischenprodukt der H2S-Oxidation von R. julia ist elementarer Schwefel, der sowohl außen als auch innen gespeichert wird. Als Schwefelquellen dienen Schwefelwasserstoff, elementarer Schwefel und die Aminosäuren Cystein und Glutathion. Sulfat wird hingegen nicht genutzt. Rhodopseudomonas julia kann Ammoniak und Casaminosäuren als Stickstoffquellen nutzen. Nitrat wird hierfür nicht genutzt. Bei geringen Sauerstoffgehalt, also unter mikroaeroben Bedingungen kann R. Julia auch chemoorganotroph wachsen. Die Art wurde aus einer säurehaltigen Schwefelquelle isoliert.
Rhodopseudomonas palustris ist in der Lage, unter Sauerstoffausschluss eine Vielzahl von verschiedenen aromatischen Verbindungen abzubauen.[3][4]
Chemotaxonomische Merkmale
[Bearbeiten | Quelltext bearbeiten]Rhodopseudomonas enthält Ubichinon mit 10 Isopreneinheiten (Q-10). Der Hauptbestandteil der zellulären Fettsäuren ist die geradkettige, einfach ungesättigte Ölsäure (C18:1).[1]
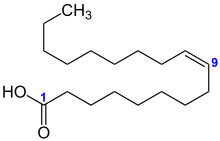
Bei Rhodopseudomonas ist in der LPS-Schicht der äußeren Membran statt dem N-Acetylglucosamin die 2,3-Diamino-2,3-didesoxyglucose enthalten. Diese Lipid A-Variante findet sich sonst nur bei wenigen Bakterien, Beispiele sind Nitrobacter und Bradyrhizobium.[5]
Rhodopseudomonas enthält das Bacteriochlorophyll a und Carotinoide vom Typ der Spirilloxanthine.[1] Die früher geführten Arten Rhodopseudomonas viridis und Rhodopseudomonas sulfoviridis enthalten zusätzlich das Bacteriochlorophyll b, sie wurden aufgrund weitere Untersuchungen von Akira Hiraishi zu der Gattung Blastochloris überführt.[6]
Ökologie
[Bearbeiten | Quelltext bearbeiten]Rhodopseudomonas kommt im Süßwasser vor und ist besonders häufig in eutrophen Gewässern anzutreffen. Es bevorzugt Wasser mit neutralen pH-Werten.[1]
Geschichte
[Bearbeiten | Quelltext bearbeiten]Die Typusart Rhodopseudomonas palustris geht auf den österreichischen Botaniker Hans Molisch zurück, der sie im Jahre 1907 erstmals beschrieb.[7] Hans Molisch beschrieb damals 2 Arten, Rhodobacillus palustris und Rhodovibrio paraus. Im Jahr 1944 wurden diese Arten von Cornelis Bernardus van Niel mit der Art Rhodomonas palustris zusammengefasst und als Rhodopseudomonas palustris bezeichnet.[8]
Die früher zur Gattung Rhodopseudomonas gestellte Art Rhodopseudomonas viridis, seit 1997 als Blastochloris viridis geführt[6], diente in den 1980er-Jahren als Modellorganismus für die Aufklärung der chemischen Abläufe der Photosynthese. Im Jahre 1985 wurde der Aufbau der beteiligten Moleküle von Hartmut Michel, Johann Deisenhofer, Robert Huber und Mitarbeitern aufgeklärt. Die drei Wissenschaftler erhielten hierfür 1988 den Nobelpreis für Chemie.[9] Von R. palustris wurde die DNA mehrere Stämme vollständig sequenziert.
Systematik
[Bearbeiten | Quelltext bearbeiten]Die Gattung Rhodopseudomonas zählt zu der Familie der Nitrobacteraceae. Zuvor wurde sie in der Familie Bradyrhizobiaceae geführt. Diese Familie wurde im Jahr 2020 aufgrund einer großangelegten Studie der Genome der Mitglieder der Klasse Alphaproteobacteria aufgelöst.[10]
Es folgt eine Liste der im Oktober 2020 geführten Arten:[11]
- Rhodopseudomonas faecalis
- Rhodopseudomonas harwoodiae
- Rhodopseudomonas julia
- Rhodopseudomonas palustris
- Rhodopseudomonas parapalustris
- Rhodopseudomonas pentothenatexigens
- Rhodopseudomonas pseudopalustris
- Rhodopseudomonas rhenobacensis
- Rhodopseudomonas telluris
- Rhodopseudomonas thermotolerans
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c d e f George M. Garrity: Bergey's manual of systematic bacteriology. 2. Auflage. Springer, New York, 2005, Vol. 2: The Proteobacteria Part C: The Alpha-, Beta-, Delta-, and Epsilonproteabacteria ISBN 0-387-24145-0
- ↑ a b Gerhard Drews: Mikrobiologisches Praktikum, 1983. ISBN 978-3-642-68747-1
- ↑ Caroline S. Harwood und Jane Gibson: Anaerobic and aerobic metabolism of diverse aromatic compounds by the photosynthetic bacterium Rhodopseudomonas palustris. In: Applied Environmental Microbiology. (1988), 54, S. 712–717
- ↑ Yasuhiro Oda, Ynte P. de Vries, Larry J. Forney und Jan C. Gottschal: Acquisition of the ability for Rhodopseudomonas palustris to degrade chlorinated benzoic acids as the sole carbon source. In: FEMS Microbiology Ecology (2001) 38, S. 133–139
- ↑ Georg Fuchs (Hrsg.): Allgemeine Mikrobiologie. 9. Auflage. Georg Thieme Verlag, Stuttgart 2014, ISBN 978-3-13-444609-8
- ↑ a b Akira Hiraishi: Transfer of the bacteriochlorophyll b-containing phototrophic bacteria Rhodopseudomonas viridis and Rhodopseudomonas sulfoviridis to the genus Blastochloris gen. nov In: International journal of systematic bacteriology (1997) 47(1): S. 217-9 doi:10.1099/00207713-47-1-217
- ↑ Hans Molisch: Die Purpurbakterien nach neuen Untersuchungen, Vol I-VII. (1907) G. Fischer, Jena, S. 1–95
- ↑ Cornelis Bernardus van Niel: The Culture, General Physiology, Morphology, and Classification of the Non-Sulfu Purple And Brown Bacteria In: Bacteriology Reviews. 1944, Band 8, Ausgabe 1, S. 1–118. PMID 16350090
- ↑ Lexikon der Biologie, Eintrag "Phototrophe Bakterien".
- ↑ Anton Hördt, Marina García López, Jan P Meier-Kolthoff, Marcel Schleuning, Lisa-Maria Weinhold, Brian J Tindall, Sabine Gronow, Nikos C. Kyrpides, Tanja Woyke und Markus Göker: Analysis of 1,000+ Type-Strain Genomes Substantially Improves Taxonomic Classification of Alphaproteobacteria. In: Front Microbiol (2020) Band 11: S. 468. doi:10.3389/fmicb.2020.00468
- ↑ Jean Euzéby, Aidan C. Parte: Rhodopseudomonas. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 10. August 2020.
Literatur
[Bearbeiten | Quelltext bearbeiten]- George M. Garrity: Bergey's manual of systematic bacteriology. 2. Auflage. Springer, New York, 2005, Vol. 2: The Proteobacteria Part C: The Alpha-, Beta-, Delta-, and Epsilonproteabacteria ISBN 0-387-24145-0
- Eugene Rosenberg, Edward F. DeLong, Stephen Lory, Erko Stackebrandt und Fabiano Thompson: The Prokaryotes. Alphaproteobacteria and Betaproteobacteria Springer, Berlin, Heidelberg 2014, ISBN 978-3-642-30197-1