Spike-Glykoprotein von SARS-CoV-2
| Spike-Glykoprotein von SARS-CoV-2 | ||
|---|---|---|
| ||
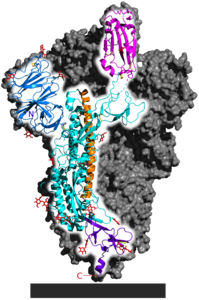
| Spike-Glykoprotein in der Präfusionsfaltung, nach PDB 6VSB (bestimmt mit Kryo-EM) | ||
| Andere Namen |
S-Glykoprotein (SARS-CoV-2) | |
| Masse/Länge Primärstruktur | 1.273 Aminosäuren, 141 kDa[1] | |
| Bezeichner | ||
| Externe IDs | ||

Das Spike-Glykoprotein von SARS-CoV-2 ist eine nach außen ragende Proteinstruktur (Peplomer) des SARS-CoV-2 Virions. Es ist ein transmembranes Glykoprotein auf der Virusoberfläche des SARS-CoV-2 und dient als Ligand zum Andocken an ACE2 auf der Zelloberfläche sowie als fusogenes Protein zum Zelleintritt. Wie bei den anderen Mitgliedern der Coronaviridae bilden die Spike-Gykoproteine die markante und namensgebende (lateinisch Corona ‚Krone‘) Oberflächenstruktur dieser Virionen.
Eigenschaften
[Bearbeiten | Quelltext bearbeiten]

Die SARS-CoV-2 Virion bindet mit Hilfe der Spike-Glykoproteine an den ACE2-Rezeptor von Körperzellen, mit denen es verschmilzt und sich darin vermehrt (Wirtszellen).[4]
Bindung und Fusion mit den Wirtszellen
[Bearbeiten | Quelltext bearbeiten]Das Spike-Glykoprotein ist ein homotrimeres Protein[5] und eines der vier strukturellen Proteine des SARS-CoV-2.[6] Es bestimmt den Wirts- und Zelltropismus des SARS-CoV-2.[6][7] Auf der Oberfläche eines Virions befinden sich 15–30 frei rotierbare Homotrimere des Spike-Glykoproteins.[8] Jedes der drei gleichen Monomere besitzt drei interne Disulfidbrücken.[5] Jede der drei gleichen Untereinheiten wird durch Proteolyse von einer Furin-artigen Protease vor der Position 686 in zwei Teile S1 und S2 gespalten, wobei die gespaltenen Teile S1 und S2 aneinander gebunden bleiben. Im Gegensatz zu anderen Coronaviren besitzt die Schnittstelle die Sequenz RRAR, welche die Spaltung erleichtert.[9] Im S1-Bereich liegt nach einem Signalpeptid zunächst die N-terminale Domäne (NTD), gefolgt von der Rezeptorbindungsdomäne (RBD) zum Andocken an die Wirtszelle.[10] Das Signalpeptid enthält 13 Aminosäuren, die spiralförmig und hydrophob (wassermeidend) angeordnet sind.[10] In der NTD liegt die Sequenz GTNGTKR, die vermutlich die Bindung an weitere Membranproteine ermöglicht.[8] N-Glykosylierungen an N165 und N234 stabilisieren die Bindung der RBD an ACE2.[2] N-Glykosylierungen an N61 und N603 führen zu einer erhöhten Zugänglichkeit der Schnittstelle für Proteasen.[8]
S2 wird nach Aufnahme ins Endosom weiter durch TMPRSS2 vor der Position 816 gespalten, wodurch S2' (Position 816 – 1273) entsteht[5] und die Fusionsdomäne aktiviert wird.[7][11] Diese Spaltung wird durch Cathepsin L nichtessentiell unterstützt.[12] Im S2-Bereich liegen das Fusionspeptid (FP) und das interne fusionspeptid (IFP) für die Verschmelzung von Virus- und Zellmembran, zwei Heptad-Repeat-Domänen (HR1 und HR2), eine Transmembrandomäne und zuletzt eine kurze C-terminale Domäne, die im Zytosol liegt.[10] Bei S2' fehlt durch die Spaltung das Fusionspeptid FP.[10] Die Proteine M und E des SARS-CoV-2 modulieren den Proteintransport neugebildeter Spike-Glykoproteine über unterschiedliche Mechanismen.[13] Die Transmembrandomäne ist die zweite hydrophobe Aminosäuresequenz des Spike-Glykoproteins.[10]
Proteinfaltungen
[Bearbeiten | Quelltext bearbeiten]
Das Spike-Glykoprotein besitzt drei mögliche Proteinfaltungen: die native Präfusionsform, die Prä-Haarnadelschleifenform und die Postfusions-Haarnadelschleifen-Form.[5] Bei der Membranfusion mit der Wirtszelle bilden Coiled-Coil-Strukturen (mit Heptadenmuster) ein Trimer von Haarnadelschleifen, wodurch das Fusionspeptid in die Nähe des C-terminalen Bereichs der extrazellulären Proteindomäne gebracht wird, woraufhin die Membranfusion mit der Membran der Wirtszelle erfolgt.[5] Durch die Bindung an den Rezeptor wird eine Änderung der Faltung induziert, welche das Fusionspeptid stärker exponiert, wodurch die Membranfusion ausgelöst wird.[16] Das Fusionspeptid besteht aus zwei Teilen an den Positionen 816 – 837 und 835 – 855.[5] Die Transmembrandomäne liegt bei den Aminosäuren 1214 – 1236.[5] Durch die Fusion wird das Virusinnere (englisch core) mit dem viralen Genom ins Zytosol freigesetzt, woraufhin die Translation der viralen Proteine beginnt.[6] Wie viele andere fusogene Proteine führen Spike-Glykoproteine von humanen Coronaviren[17] und auch das von SARS-CoV-2 zur Bildung von Syncytien,[18][19] vermutlich zur Infektion von Nachbarzellen.[10]
Manche SARS-CoV-2-Impfstoffe verwenden als Antigen eine Variante des S-Glykoproteins, die zwei geänderte Proline in der Nähe der Fusionsdomäne aufweist, welche die Proteinfaltung vor der Membranfusion stabilisieren (engl. 2P-prefusion-stabilised). Bei der 2P-Variante wurden zwei Aminosäuren gegen Proline getauscht: an Position 1060 war zuvor ein Valin, an Position 1061 war zuvor ein Leucin.[20] Die 2P-Variante wurde für Coronaviren erstmals beim MERS-CoV beschrieben.[20] Die Analogie der 2P-Variante bei SARS-CoV-2 wurde bestätigt.[21]
Immunologie
[Bearbeiten | Quelltext bearbeiten]

Antikörper gegen das Spike-Glykoprotein werden sowohl nach Infektion mit SARS-CoV-2 als auch nach Impfung mit COVID-19-Impfstoffen gebildet. Neutralisierende Antikörper gegen SARS-CoV-2 binden zu einem Anteil von etwa 90 % an die RBD im S1.[16][11] Die monoklonalen Antikörper Bamlanivimab, Casirivimab, Cilgavimab, Etesevimab, Imdevimab, Regdanvimab, Sotrovimab und Tixagevimab binden an die Rezeptorbindungsdomäne (RBD) des Spike-Glykoproteins von SARS-CoV-2. Viele Epitope auf der Oberfläche des Spike-Glykoproteins sind durch umfangreiche Glykosylierungen maskiert.[2] Da die RBD im Vergleich zu anderen Oberflächenstrukturen weniger durch Glykosylierungen maskiert ist, wirkt die RBD vermutlich für neutralisierende Antikörper immundominant.[22] Antikörper-Fluchtmutationen treten vor allem in einem bestimmten Bereich der RBD auf, was ebenfalls auf die Immundominanz der RBD für Antikörper hinweist.[23] Neutralisierende Antikörper gegen das Spike-Glykoprotein werden nach ihren Bindungseigenschaften in 4 Klassen eingeteilt:[24]
- ACE2-blockierende Antikörper, welche die offene Faltungsform des Spike-Glykoproteins binden (Klasse 1)
- ACE2-blockierende Antikörper, welche die offene und die geschlossene Faltungsform des Spike-Glykoproteins binden (Klasse 2)
- Nicht-ACE2-blockierende Antikörper, welche die offene und die geschlossenen Faltungsform der RBD des Spike-Glykoproteins binden (Klasse 3)
- Neutralisierende Antikörper, welche außerhalb der ACE2-Bindungsstelle und nur an die offene Faltungsform binden (Klasse 4)
Die wenigen neutralisierenden Antikörper, welche nicht an die RBD binden, binden meist an die NTD.[16] Sie binden gehäuft an die Positionen 14–20, 140–158 und 245–264.[25]
Es gibt in Mäusen eine Kreuzreaktivität von neutralisierenden Antikörpern gegen das S-Glykoprotein, die sowohl den Zelleintritt von SARS-CoV als auch von SARS-CoV-2 hemmen.[26] Beide SARS-assoziierten Coronaviren (1 und 2) verwenden den gleichen Rezeptor zum Zelleintritt, das Angiotensin-konvertierende Enzym 2 (ACE2), während MERS-CoV die Dipeptidylpeptidase 4 (CD26) verwendet.[27] Die Bindungsfläche zu ACE2 unterscheidet sich zwischen den RBD der Spike-Glykoproteine von SARS-CoV-1 und -2, weshalb nicht alle ihrer neutralisierenden Antikörper kreuzreaktiv sind.[10] S1 ist variabler als S2 in der Aminosäuresequenz, weshalb eher S2-bindende Antikörper kreuzreaktiv sind.[10]
Im Spike-Glykoprotein des SARS-CoV-2 wurden für den Menschen 13 Epitope für MHC I (erzeugen eine zelluläre Immunantwort) und 3 für MHC II (erzeugen eine humorale Immunantwort) identifiziert.[28] Konservierte Epitope wurden im S-Glykoprotein und im Nukleokapsidprotein identifiziert, die sich für breitenwirksame Impfstoffe eignen könnten.[29]
Mutationen
[Bearbeiten | Quelltext bearbeiten]Mutationen des Spike-Glykoproteins von SARS-CoV-2 entstehen bei der Replikation des viralen Genoms. Diese sind überwiegend Mutationen ohne oder mit geringen Fitnesskosten.[16] Daneben werden auch Mutationen beobachtet, die sich stark auf die Aminosäuresequenz des Spike-Glykoproteins auswirken.[16] Diese wirken sich üblicherweise auf die Pathogenität, Infektivität, Übertragbarkeit und/oder Antigenität aus.[16] Einige der Mutationen mit stärkerer Auswirkung sind im Laufe der Covid-19-Pandemie mehrfach und unabhängig voneinander in der Sequenz des Spike-Glykoproteins entstanden.[16]
An der Position 614 – in der Rezeptorbindungsdomäne (RBD) – treten gehäuft nichtsynonyme Mutationen auf, was auf eine stärkere positive Selektion für diese Stelle im Spike-Glykoprotein hindeutet.[16] Die Mutationen D614G, N501Y, E484K-Q, K417N/T und L452R sind mit erhöhter Virusproduktion, erhöhter Übertragbarkeit, geänderter Virulenz und vermutlich auch mit Immunflucht assoziiert.[30][31] Die Mutation D614G vermittelt eine höhere Infektivität und Übertragbarkeit des SARS-CoV-2.[16][32][33] Die Mutation N439K führt zu einer höheren Affinität zum Rezeptor und mindert die Bindung mancher neutralisierender Antikörper.[16] Die Mutation Y453F führt ebenso zu einer größeren Affinität für ACE2.[16] Mutationen an der Position 484 führen zu einer geminderten Bindung durch neutralisierende Antikörper und sind somit Fluchtmutationen,[34] vor allem E484K, E484Q, E484P, E484A, E484D und E484G.[16][35] Ebenso an der Position die Mutationen K444R, K444N, K444Q, V445E und an Position 477 die Mutationen S477G, S477N and S477R.[16] Als weitere Antikörper-Fluchtmutationen an anderen Positionen wurden K417N/T, L452R, Y453F, S477N, und N501Y beschrieben.[36][37][33]
In der NTD wurden die Bereiche 140–156 und 246–260 als Orte für Fluchtmutationen beschrieben, darunter N148S, K150R, K150E, K150T, K150Q und S151P.[16] Die Deletion der Aminosäuren an der Position 69 und 70 – in der NTD – erhöht die Infektivität.[16] In der Omikron-Variante besitzen die Mutationen 478K, E484A, Q493R und N501Y den stärksten Einfluss unter ihren über 30 geänderten Aminosäuren im Spike-Glykoprotein.[38]
Auswirkungen
[Bearbeiten | Quelltext bearbeiten]Das Spike-Glykoprotein trägt zur Pathogenese der Covid-19 Krankheit durch verschiedene Mechanismen bei:
- Die Bindung zum ACE2 Rezeptor kann zur Deregulierung der Funktionen dieses Enzyms in mehreren Organen führen, darunter in den Lungen, den Gefäßen, dem Herzen, den Nieren, dem Darm und dem Gehirn. Viele Symptome und Folgen von Covid-19 können durch diese Deregulierung verstanden werden.[39]
- Unter anderen Mechanismen, die zur Deregulierung der Blutgerinnungsmechanismen in den Gefäßen durch SARS-CoV-2 führen können, steht auch der direkte oder indirekte Einfluss des S1-Bereichs des Spike-Glykoproteins auf Rezeptoren und andere Strukturen der Blutgefäße.[40][41]
- Das Spike-Glykoprotein kann sich auch zu anderen Rezeptoren binden und dadurch zur Deregulierung weiterer Funktionen führen.[42]
Verwendung
[Bearbeiten | Quelltext bearbeiten]In Impfstoffen
[Bearbeiten | Quelltext bearbeiten]Alle zugelassenen Impfstoffe enthalten als Antigen das Spike-Glykoprotein oder sein Gen. Oftmals wird die 2P-Variante verwendet, bei der die Proteinfaltung vor der Fusion des Virus mit der Zellmembran stabilisiert wurde. Dies soll dazu dienen, „das theoretische Risiko der Erzeugung infektionsverstärkender Antikörper zu reduzieren“.[43]
In Neutralisation-Assays
[Bearbeiten | Quelltext bearbeiten]Zur Untersuchung von Blutproben auf ihre Neutralisierungsfähigkeit von SARS-CoV-2 werden aufgrund der Gefährlichkeit des Virus häufig statt der eigentlichen Virionen nur pseudotypisierte Viren eingesetzt, dies sind modifizierte („chimäre“) Viren, die das Spike-Glykoprotein von SARS-CoV-2 enthalten.[44][45][46] Es werden hierbei lentivirale Vektoren verwendet, die auch Spikeproteine an der Oberfläche haben, wobei der genetische Code für die (Lentivirus-)Spike-Glykoproteine durch den Code des SARS-CoV-2-Spike ausgetauscht ist.
Literatur
[Bearbeiten | Quelltext bearbeiten]- Zunlong Ke, Joaquin Oton u. a.: Structures and distributions of SARS-CoV-2 spike proteins on intact virions. In: Nature. 588, 2020, S. 498, doi:10.1038/s41586-020-2665-2.
- Bálint Kiss, Zoltán Kis, Bernadett Pályi, Miklós S. Z. Kellermayer: Topography, Spike Dynamics, and Nanomechanics of Individual Native SARS-CoV-2 Virions. In: Nano Letters. 21, 2021, S. 2675, doi:10.1021/acs.nanolett.0c04465.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ surface glycoprotein (Severe acute respiratory syndrome coronavirus 2) (NIH-NLM Datenbank)
- ↑ a b c L. Casalino, Z. Gaieb, J. A. Goldsmith, C. K. Hjorth, A. C. Dommer, A. M. Harbison, C. A. Fogarty, E. P. Barros, B. C. Taylor, J. S. McLellan, E. Fadda, R. E. Amaro: Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein. In: ACS central science. Band 6, Nummer 10, Oktober 2020, S. 1722–1734, doi:10.1021/acscentsci.0c01056, PMID 33140034, PMC 7523240 (freier Volltext).
- ↑ Franz X. Heinz, Karin Stiasny: Profile of SARS-CoV-2. In: Wiener klinische Wochenschrift. Band 132. Springer Austria, 30. Oktober 2020, ISSN 0043-5325, S. 635–644; hier: 637, doi:10.1007/s00508-020-01763-1, PMID 33125580, PMC 7597426 (freier Volltext) – (springer.com [PDF; 1,1 MB; abgerufen am 17. Dezember 2021]).
- ↑ Mechanisms of SARS-CoV-2 entry into cells (www.nature.com/, 5. Oktober 2021)
- ↑ a b c d e f g UniProt S - Spike glycoprotein - Severe acute respiratory syndrome coronavirus 2 (2019-nCoV) - S gene & protein. Abgerufen am 3. April 2022.
- ↑ a b c Y. Wang, M. Grunewald, S. Perlman: Coronaviruses: An Updated Overview of Their Replication and Pathogenesis. In: Methods in molecular biology. Band 2203, 2020, S. 1–29, doi:10.1007/978-1-0716-0900-2_1, PMID 32833200, PMC 7682345 (freier Volltext).
- ↑ a b C. Zhu, G. He, Q. Yin, L. Zeng, X. Ye, Y. Shi, W. Xu: Molecular biology of the SARs-CoV-2 spike protein: A review of current knowledge. In: Journal of medical virology. Band 93, Nummer 10, 10 2021, S. 5729–5741, doi:10.1002/jmv.27132, PMID 34125455, PMC 8427004 (freier Volltext).
- ↑ a b c S. B. Kadam, G. S. Sukhramani, P. Bishnoi, A. A. Pable, V. T. Barvkar: SARS-CoV-2, the pandemic coronavirus: Molecular and structural insights. In: Journal of basic microbiology. Band 61, Nummer 3, März 2021, S. 180–202, doi:10.1002/jobm.202000537, PMID 33460172, PMC 8013332 (freier Volltext).
- ↑ R. Kumavath, D. Barh, B. S. Andrade, M. Imchen, F. F. Aburjaile, A. Ch, D. L. Rodrigues, S. Tiwari, K. J. Alzahrani, A. Góes-Neto, M. E. Weener, P. Ghosh, V. Azevedo: The Spike of SARS-CoV-2: Uniqueness and Applications. In: Frontiers in immunology. Band 12, 2021, S. 663912, doi:10.3389/fimmu.2021.663912, PMID 34305894, PMC 8297464 (freier Volltext).
- ↑ a b c d e f g h X. Xia: Domains and Functions of Spike Protein in Sars-Cov-2 in the Context of Vaccine Design. In: Viruses. Band 13, Nummer 1, Januar 2021, S. , doi:10.3390/v13010109, PMID 33466921, PMC 7829931 (freier Volltext).
- ↑ a b P. V'kovski, A. Kratzel, S. Steiner, H. Stalder, V. Thiel: Coronavirus biology and replication: implications for SARS-CoV-2. In: Nature reviews. Microbiology. Band 19, Nummer 3, 03 2021, S. 155–170, doi:10.1038/s41579-020-00468-6, PMID 33116300, PMC 7592455 (freier Volltext).
- ↑ C. B. Jackson, M. Farzan, B. Chen, H. Choe: Mechanisms of SARS-CoV-2 entry into cells. In: Nature reviews. Molecular cell biology. Band 23, Nummer 1, 01 2022, S. 3–20, doi:10.1038/s41580-021-00418-x, PMID 34611326, PMC 8491763 (freier Volltext).
- ↑ B. Boson, V. Legros, B. Zhou, E. Siret, C. Mathieu, F. L. Cosset, D. Lavillette, S. Denolly: The SARS-CoV-2 envelope and membrane proteins modulate maturation and retention of the spike protein, allowing assembly of virus-like particles. In: Journal of Biological Chemistry. Band 296, 2021 Jan-Jun, S. 100111, doi:10.1074/jbc.RA120.016175, PMID 33229438, PMC 7833635 (freier Volltext).
- ↑ A. C. Walls, X. Xiong, Y. J. Park, M. A. Tortorici, J. Snijder, J. Quispe, E. Cameroni, R. Gopal, M. Dai, A. Lanzavecchia, M. Zambon, F. A. Rey, D. Corti, D. Veesler: Unexpected Receptor Functional Mimicry Elucidates Activation of Coronavirus Fusion. In: Cell. Band 176, Nummer 5, 02 2019, S. 1026–1039.e15, doi:10.1016/j.cell.2018.12.028, PMID 30712865, PMC 6751136 (freier Volltext).
- ↑ X. Fan, D. Cao, L. Kong, X. Zhang: Cryo-EM analysis of the post-fusion structure of the SARS-CoV spike glycoprotein. In: Nature Communications. Band 11, Nummer 1, 07 2020, S. 3618, doi:10.1038/s41467-020-17371-6, PMID 32681106, PMC 7367865 (freier Volltext).
- ↑ a b c d e f g h i j k l m n o W. T. Harvey, A. M. Carabelli, B. Jackson, R. K. Gupta, E. C. Thomson, E. M. Harrison, C. Ludden, R. Reeve, A. Rambaut,. , S. J. Peacock, D. L. Rober: SARS-CoV-2 variants, spike mutations and immune escape. In: Nature reviews. Microbiology. Band 19, Nummer 7, 07 2021, S. 409–424, doi:10.1038/s41579-021-00573-0, PMID 34075212, PMC 8167834 (freier Volltext).
- ↑ S. Belouzard, J. K. Millet, B. N. Licitra, G. R. Whittaker: Mechanisms of coronavirus cell entry mediated by the viral spike protein. In: Viruses. Band 4, Nummer 6, 06 2012, S. 1011–1033, doi:10.3390/v4061011, PMID 22816037, PMC 3397359 (freier Volltext).
- ↑ J. Buchrieser, J. Dufloo, M. Hubert, B. Monel, D. Planas, M. M. Rajah, C. Planchais, F. Porrot, F. Guivel-Benhassine, S. Van der Werf, N. Casartelli, H. Mouquet, T. Bruel, O. Schwartz: Syncytia formation by SARS-CoV-2-infected cells. In: The EMBO Journal. Band 39, Nummer 23, 12 2020, S. e106267, doi:10.15252/embj.2020106267, PMID 33051876, PMC 7646020 (freier Volltext).
- ↑ Z. Zhang, Y. Zheng, Z. Niu, B. Zhang, C. Wang, X. Yao, H. Peng, D. N. Franca, Y. Wang, Y. Zhu, Y. Su, M. Tang, X. Jiang, H. Ren, M. He, Y. Wang, L. Gao, P. Zhao, H. Shi, Z. Chen, X. Wang, M. Piacentini, X. Bian, G. Melino, L. Liu, H. Huang, Q. Sun: SARS-CoV-2 spike protein dictates syncytium-mediated lymphocyte elimination. In: Cell death and differentiation. Band 28, Nummer 9, 09 2021, S. 2765–2777, doi:10.1038/s41418-021-00782-3, PMID 33879858, PMC 8056997 (freier Volltext).
- ↑ a b J. Pallesen, N. Wang, K. S. Corbett, D. Wrapp, R. N. Kirchdoerfer, H. L. Turner, C. A. Cottrell, M. M. Becker, L. Wang, W. Shi, W. P. Kong, E. L. Andres, A. N. Kettenbach, M. R. Denison, J. D. Chappell, B. S. Graham, A. B. Ward, J. S. McLellan: Immunogenicity and structures of a rationally designed prefusion MERS-CoV spike antigen. In: Proceedings of the National Academy of Sciences. Band 114, Nummer 35, 08 2017, S. E7348–E7357, doi:10.1073/pnas.1707304114, PMID 28807998, PMC 5584442 (freier Volltext).
- ↑ D. Wrapp, N. Wang, K. S. Corbett, J. A. Goldsmith, C. L. Hsieh, O. Abiona, B. S. Graham, J. S. McLellan: Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. In: Science. Band 367, Nummer 6483, 03 2020, S. 1260–1263, doi:10.1126/science.abb2507, PMID 32075877, PMC 7164637 (freier Volltext).
- ↑ Y. Watanabe, Z. T. Berndsen, J. Raghwani, G. E. Seabright, J. D. Allen, O. G. Pybus, J. S. McLellan, I. A. Wilson, T. A. Bowden, A. B. Ward, M. Crispin: Vulnerabilities in coronavirus glycan shields despite extensive glycosylation. In: Nature Communications. Band 11, Nummer 1, 05 2020, S. 2688, doi:10.1038/s41467-020-16567-0, PMID 32461612, PMC 7253482 (freier Volltext).
- ↑ A. J. Greaney, A. N. Loes, K. H. Crawford, T. N. Starr, K. D. Malone, H. Y. Chu, J. D. Bloom: Comprehensive mapping of mutations in the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human plasma antibodies. In: Cell host & microbe. Band 29, Nummer 3, 03 2021, S. 463–476.e6, doi:10.1016/j.chom.2021.02.003, PMID 33592168, PMC 7869748 (freier Volltext).
- ↑ C. O. Barnes, C. A. Jette, M. E. Abernathy, K. A. Dam, S. R. Esswein, H. B. Gristick, A. G. Malyutin, N. G. Sharaf, K. E. Huey-Tubman, Y. E. Lee, D. F. Robbiani, M. C. Nussenzweig, A. P. West, P. J. Bjorkman: SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. In: Nature. Band 588, Nummer 7839, 12 2020, S. 682–687, doi:10.1038/s41586-020-2852-1, PMID 33045718, PMC 8092461 (freier Volltext).
- ↑ M. McCallum, A. De Marco, F. A. Lempp, M. A. Tortorici, D. Pinto, A. C. Walls, M. Beltramello, A. Chen, Z. Liu, F. Zatta, S. Zepeda, J. di Iulio, J. E. Bowen, M. Montiel-Ruiz, J. Zhou, L. E. Rosen, S. Bianchi, B. Guarino, C. S. Fregni, R. Abdelnabi, S. C. Foo, P. W. Rothlauf, L. M. Bloyet, F. Benigni, E. Cameroni, J. Neyts, A. Riva, G. Snell, A. Telenti, S. P. Whelan, H. W. Virgin, D. Corti, M. S. Pizzuto, D. Veesler: N-terminal domain antigenic mapping reveals a site of vulnerability for SARS-CoV-2. In: Cell. Band 184, Nummer 9, 04 2021, S. 2332–2347.e16, doi:10.1016/j.cell.2021.03.028, PMID 33761326, PMC 7962585 (freier Volltext).
- ↑ A. C. Walls, Y. J. Park, M. A. Tortorici, A. Wall, A. T. McGuire, D. Veesler: Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. In: Cell. [elektronische Veröffentlichung vor dem Druck] März 2020, doi:10.1016/j.cell.2020.02.058, PMID 32155444.
- ↑ E. Prompetchara, C. Ketloy, T. Palaga: Immune responses in COVID-19 and potential vaccines: Lessons learned from SARS and MERS epidemic. In: Asian Pacific journal of allergy and immunology. [elektronische Veröffentlichung vor dem Druck] März 2020, doi:10.12932/AP-200220-0772, PMID 32105090.
- ↑ M. Bhattacharya, A. R. Sharma, P. Patra, P. Ghosh, G. Sharma, B. C. Patra, S. S. Lee, C. Chakraborty: Development of epitope-based peptide vaccine against novel coronavirus 2019 (SARS-COV-2): Immunoinformatics approach. In: Journal of medical virology. [elektronische Veröffentlichung vor dem Druck] Februar 2020, doi:10.1002/jmv.25736, PMID 32108359.
- ↑ S. F. Ahmed, A. A. Quadeer, M. R. McKay: Preliminary Identification of Potential Vaccine Targets for the COVID-19 Coronavirus (SARS-CoV-2) Based on SARS-CoV Immunological Studies. In: Viruses. Band 12, Nummer 3, Februar 2020, S. , doi:10.3390/v12030254, PMID 32106567.
- ↑ V. Papanikolaou, A. Chrysovergis, V. Ragos, E. Tsiambas, S. Katsinis, A. Manoli, S. Papouliakos, D. Roukas, S. Mastronikolis, D. Peschos, A. Batistatou, E. Kyrodimos, N. Mastronikolis: From delta to Omicron: S1-RBD/S2 mutation/deletion equilibrium in SARS-CoV-2 defined variants. In: Gene. Band 814, März 2022, S. 146134, doi:10.1016/j.gene.2021.146134, PMID 34990799, PMC 8725615 (freier Volltext).
- ↑ B. Bakhshandeh, Z. Jahanafrooz, A. Abbasi, M. B. Goli, M. Sadeghi, M. S. Mottaqi, M. Zamani: Mutations in SARS-CoV-2; Consequences in structure, function, and pathogenicity of the virus. In: Microbial Pathogenesis. Band 154, Mai 2021, S. 104831, doi:10.1016/j.micpath.2021.104831, PMID 33727169, PMC 7955574 (freier Volltext).
- ↑ D. C. Groves, S. L. Rowland-Jones, A. Angyal: The D614G mutations in the SARS-CoV-2 spike protein: Implications for viral infectivity, disease severity and vaccine design. In: Biochemical and biophysical research communications. Band 538, 01 2021, S. 104–107, doi:10.1016/j.bbrc.2020.10.109, PMID 33199022, PMC 7643658 (freier Volltext).
- ↑ a b C. B. Jackson, L. Zhang, M. Farzan, H. Choe: Functional importance of the D614G mutation in the SARS-CoV-2 spike protein. In: Biochemical and biophysical research communications. Band 538, 01 2021, S. 108–115, doi:10.1016/j.bbrc.2020.11.026, PMID 33220921, PMC 7664360 (freier Volltext).
- ↑ I. Lazarevic, V. Pravica, D. Miljanovic, M. Cupic: Immune Evasion of SARS-CoV-2 Emerging Variants: What Have We Learnt So Far? In: Viruses. Band 13, Nummer 7, 06 2021, S. , doi:10.3390/v13071192, PMID 34206453, PMC 8310325 (freier Volltext).
- ↑ E. Boehm, I. Kronig, R. A. Neher, I. Eckerle, P. Vetter, L. Kaiser: Novel SARS-CoV-2 variants: the pandemics within the pandemic. In: Clinical microbiology and infection : the official publication of the European Society of Clinical Microbiology and Infectious Diseases. Band 27, Nummer 8, August 2021, S. 1109–1117, doi:10.1016/j.cmi.2021.05.022, PMID 34015535, PMC 8127517 (freier Volltext).
- ↑ D. Focosi, F. Maggi: Neutralising antibody escape of SARS-CoV-2 spike protein: Risk assessment for antibody-based Covid-19 therapeutics and vaccines. In: Reviews in Medical Virology. Band 31, Nummer 6, 11 2021, S. e2231, doi:10.1002/rmv.2231, PMID 33724631, PMC 8250244 (freier Volltext).
- ↑ A. Winger, T. Caspari: The Spike of Concern-The Novel Variants of SARS-CoV-2. In: Viruses. Band 13, Nummer 6, 05 2021, S. , doi:10.3390/v13061002, PMID 34071984, PMC 8229995 (freier Volltext).
- ↑ S. Kannan, P. Shaik Syed Ali, A. Sheeza: Omicron (B.1.1.529) - variant of concern - molecular profile and epidemiology: a mini review. In: European review for medical and pharmacological sciences. Band 25, Nummer 24, 12 2021, S. 8019–8022, doi:10.26355/eurrev_202112_27653, PMID 34982466.
- ↑ Ashraf UM, Abokor AA, Edwards JM, Waigi EW, Royfman RS, Hasan SA, Smedlund KB, Hardy AMG, Chakravarti R, Koch LG. SARS-CoV-2, ACE2 expression, and systemic organ invasion. Physiol Genomics, doi:10.1152/physiolgenomics.00087.2020, PMID 33275540, PMC 7900915 (freier Volltext).
- ↑ Farooqui AA, Farooqui T, Sun GY, Lin TN, Teh DBL, Ong WY. COVID-19, Blood Lipid Changes, and Thrombosis. Biomedicines. 2023 Apr 15;11(4):1181, doi:10.3390/biomedicines11041181, PMID 37189799, PMC 10135929 (freier Volltext).
- ↑ Conway EM, Mackman N, Warren RQ, Wolberg AS, Mosnier LO, Campbell RA, Gralinski LE, Rondina MT, van de Veerdonk FL, Hoffmeister KM, Griffin JH, Nugent D, Moon K, Morrissey JH. Understanding COVID-19-associated coagulopathy. Nat Rev Immunol. 2022 Oct;22(10):639-649, doi:10.1038/s41577-022-00762-9, PMID 35931818, PMC 9362465 (freier Volltext).
- ↑ Gusev E, Sarapultsev A, Solomatina L, Chereshnev V. SARS-CoV-2-Specific Immune Response and the Pathogenesis of COVID-19. Int J Mol Sci. 2022 Feb 2;23(3):1716, doi:10.3390/ijms23031716, PMID 35163638, PMC 8835786 (freier Volltext).
- ↑ Antikörper COVID-19-Geimpfter erkennen andere Strukturen des Spike-Proteins als Antikörper SARS-CoV-2-Infizierter (Pressemitteilung des Paul-Ehrlich-Instituts 26/2021, aktualisiert 9. Dezember 2021, www.pei.de)
- ↑ J. M. Condor Capcha, G. Lambert, D. M. Dykxhoorn, A. G. Salerno, J. M. Hare, M. A. Whitt, S. Pahwa, D. T. Jayaweera, L. A. Shehadeh: Generation of SARS-CoV-2 Spike Pseudotyped Virus for Viral Entry and Neutralization Assays: A 1-Week Protocol. In: Frontiers in cardiovascular medicine. Band 7, 2020, S. 618651, doi:10.3389/fcvm.2020.618651, PMID 33521067, PMC 7843445 (freier Volltext).
- ↑ Effective screening of SARS-CoV-2 neutralizing antibodies in patient serum using lentivirus particles pseudotyped with SARS-CoV-2 spike glycoprotein (www.nature.com, 5. November 2020)
- ↑ Berthold: Was ist ein Pseudovirus Neutralization Assay? Abgerufen am 27. Januar 2021.