Benutzer:Dirk123456/Baustellenbaustelle 001/Baustelle-2/Baustelle-2.6
Abbildungen für DNA-Methylierung - Hemimethylierung und Korrektur nach Replikation
Das Ziel dieser Baustelle bestand darin, „Abbildungsserien“ aus Textzeilen mit methylierten DNA-Sequenzen durch „echte“ Bilder zu ersetzen, da die Wikipedia-Texte durch Leerzeichen „formatiert“ wurden. Dadurch sind sie beim Editieren ständig verrutsch sind. Es wurden sechs Abbildungen erstellt (Replicat-mCpG step[1-6].png) und bei Wikimedia Commons hoch geladen. Eine hier erstellte neue Version des Abschnitts hat die bisherige Version am 5. Nov. 2018 (06:30) ersetzt.
| 2004-07-10 | Erstellung des Artikels DNA-Methylierung; |
| 2005-11-25 | Erste Änderung durch Dirk123456; |
| 2006-01-27 | Komplette Überarbeitung - enthielt erstmalig den Abschnitt „DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese“ mit den Textabbildungen; |
| 2018-11-04 | Letzte Korrektur der Textausrichtung; |
| 2018-11-05 | Ersatz der Textabbildungen durch PNG-Dateien; |
- (Aktuell | Vorherige) 17:41, 10. Jul. 2004 Darkone (Diskussion | Beiträge) . . (1.182 Bytes) (+1.182) . . (danken)
- (Aktuell | Vorherige) 19:21, 25. Nov. 2005 Dirk123456 (Diskussion | Beiträge) . . (3.484 Bytes) (+2.237) . . (erweitert) (rückgängig)
- (Aktuell | Vorherige) 17:20, 27. Jan. 2006 Dirk123456 (Diskussion | Beiträge) . . (14.205 Bytes) (+10.110) . . (komplett überarbeitet) (rückgängig
- (Aktuell | Vorherige) 17:56, 4. Nov. 2018 Dirk123456 (Diskussion | Beiträge) . . (79.853 Bytes) (-1) . . (→DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese:verrutschte Methylgruppen in der Textabbildung, Schritt 4) (rückgängig) [automatisch gesichtet]
- (Aktuell | Vorherige) 06:30, 5. Nov. 2018 Dirk123456 (Diskussion | Beiträge) . . (79.908 Bytes) (+55) . . (→DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese: Austausch von "Textabbildungen" gegen "echte" Abbildungen) (kommentarlos zurücksetzen | rückgängig) [automatisch gesichtet]
Zitate[Bearbeiten | Quelltext bearbeiten]
Die Zitate dienen vor allem dazu, die Schreibweisen für Methylierungen in Sequenzen zu vergleichen und eine "übliche" zu finden. Vor allen geht es um richtig bzw. fehlgepaarte Basenpaare und um nicht-, halb- und vollständig methylierte CpG-Dinukleotide bzw. -Stellen.
Epigenetic Enhancement of the Post-replicative DNA Mismatch Repair of Mammalian Genomes by a Hemi-mCpG-Np95-Dnmt1 Axis[Bearbeiten | Quelltext bearbeiten]
| Wang et al. 2016 doi:10.1038/srep37490. PMC 5122852 (freier Volltext). PMID 27886214.</ref> | [1] |
Evolutionary Changes in Vertebrate Genome Signatures with Special Focus on Coelacanth[Bearbeiten | Quelltext bearbeiten]
| Iwasaki et al. 2014 doi:10.1093/dnares/dsu012, PMID 24800745, PMC 4195492 (freier Volltext). | [2] |
gelesen wo: Suche nach "cg suppression" or "cpg suppression"
Methylated Cytosines Mutate to Transcription Factor Binding Sites that Drive Tetrapod Evolution[Bearbeiten | Quelltext bearbeiten]
| He et al. 2015 doi:10.1093/gbe/evv205, PMID 26507798, PMC 4994754 (freier Volltext) | [3] |
DNA methylation presents distinct binding sites for human transcription factors[Bearbeiten | Quelltext bearbeiten]
| Hu et al. 2013 doi:10.7554/eLife.00726, PMID 24015356, PMC 3762332 (freier Volltext) | [4] |
Schreibweisen[Bearbeiten | Quelltext bearbeiten]
Wang et al. 2016 doi:10.1038/srep37490. PMC 5122852 (freier Volltext). PMID 27886214.
Hemi-mCpG-Np95-Dnmt1; DNA methylation at C of CpG dyads (mCpG) in vertebrate genomes is essential...; genomic mCpG; hemi-mCpG-Np95(Uhrf1)-Dnmt1 axis; hemi-methylated CpG sites; 5-mC; mCpG percentages;
E. coli that bears GmATC sequences methylated by DNA-(N6-adenine) methyltransferase (Dam)
--> keine Motive, immer nur Einzelstrang
Iwasaki et al. 2014 doi:10.1093/dnares/dsu012, PMID 24800745, PMC 4195492 (freier Volltext).
...e.g. under-representation of GTCA+TGAC in coelacanth and tetrapods...; The CG/GC ratio (often called the CpG/GpC ratio) has long been used to examine CG suppression for individual species, because this ratio is not affected by the genome G+C% difference. ...that CA/AC and TG/GT ratios.
--> Methylierte Motive werden nicht angegeben; ACHTUNG!! CpG/GpC ist das Verhältnis von CpG zu GpC! Motive werden durch + ausgedrückt, LAGER+REGAL-ähnlich, 5'-GTCA-3'+5'-TGAC-3'. Die Darstellung „mCG+GC“ hieße also nicht halbmethyliertes CpG, sondern die Gesamtheit von mCpG- und GpC-Dinukleotiden in beiden Einzelsträngen!
He et al. 2015 doi:10.1093/gbe/evv205, PMID 26507798, PMC 4994754 (freier Volltext)
...which also contains 5mC (Makapedua et al. 2011) and has fewer CGs than expected (Iwasaki et al. 2014) suggesting that at this time the efficient repair of T•G mismatch to a C•G base pair was lost... ; ....deamination product of 5mCG, TG, is the most abundant dinucleotide in vertebrates... ; three sequences CGAGTCA, 5mCGAGTCA, and TGAGTCA. We find that the deamination product, TGAC/GTCA, is better bound by the c-Jun|c-Fos heterodimer compared with 5mCGAGTCA, which in turn... ; These included discontinuous (gapped) 8-mers separated by 1- to 30-bp gaps (NNNN-(1-30)NNNN, where “-”s indicate a gap), and 8-mers of the form N-NNN—NNN-N, which are bound by hormone binding proteins (He et al. 2013). ; The longer TG-dinucleotide containing motifs that are enriched in TS-DHSs in both human and mouse genomes include those for nuclear factor 1 (NF1, TGGC—–GCCA) (de Vries et al. 1987; Blomquist et al. 1999; Whittle et al. 2009), and the canonical GR motif (G-ACA—TGT-C) (Yamamoto 1985; Beato 1989) (supplementary figs. S11 and Supplementary Data, Supplementary Material online).
--> Es wird nicht ein einziges Mal CpG hingeschrieben. Nach dem dortigen Muster wären A•T und A•C das Richtige für die Basen(fehl)paarungen und 5mCG—GC für hemimethyliertes CpG sowie 5mCG—5mCG für vollständig methyliertes CpG. Die fetten Hervorhebungen beziehen sich nicht auf die Haupt-Consensi der Motive, sondern auf die Untersuchung (CG wird zu TG und CA).
Hu et al. 2013 doi:10.7554/eLife.00726, PMID 24015356, PMC 3762332 (freier Volltext)
GTmCpGCC ; ...Arg458 and methyl group on the cytosine in one strand (5mCA). Red balls represent water molecules. (B) Asp460 further stabilizes binding to 5 mC on the other strand (5mCB) via a CH•••O (H2O—5mC) H-bond co... ;
--> konsequente Anwendung von CpG und mCpG in Motiven. Einzelne C's immer als 5mC, nie nur mC. Unterscheidung auf den beiden Strängen in einer Abbildung 5mCA und 5mCB.
Fazit[Bearbeiten | Quelltext bearbeiten]
Die Schreibweise 5mCpG gibts nicht, entweder 5mCG oder mCpG. Die Motiv-Schreibweise 5mCG—5mCG bzw. 5mCG—CG könnte man für die Abbildungen nehmen. Besser wäre wahrscheinlich dann schon „Hemi-mCpG“ oder „vollständig methylierte CpG-Stelle“ und „hemimetylierte CpG-Stelle“. Das ist aber zu lang.
Ab unverfänglichsten ist es, das alles wegzulassen und im Text daneben zu schreiben. Am schwierigsten sind Schrägstriche („/“), da sie für Basenpaare und Mehrdeutigkeiten verwendet werden.
Neue Version[Bearbeiten | Quelltext bearbeiten]
Der folgende Abschnitt wurde im Artikel DNA-Methylierung gegen den Abschnitt "Bisherige Version" ausgetauscht (ersetzt 17:56, 4. Nov. 2018).
- (Aktuell | Vorherige) 06:30, 5. Nov. 2018 Dirk123456 (Diskussion | Beiträge) . . (79.908 Bytes) (+55) . . (→DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese: Austausch von "Textabbildungen" gegen "echte" Abbildungen) (kommentarlos zurücksetzen | rückgängig) [automatisch gesichtet]
DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese[Bearbeiten | Quelltext bearbeiten]
Die identische Verdopplung der Desoxyribonukleinsäure (DNA-Replikation) ist eine wesentliche Voraussetzung für die Zellteilung und damit für die Vermehrung. Die DNA-Replikation wird dadurch gewährleistet, dass Enzyme (die DNA-abhängigen DNA-Polymerasen) den bereits vorhandenen alten Strang „lesen“ und dabei den neuen Strang „schreiben“. Dabei können Fehler auftreten. Die Fehlerstellen können durch die DNA-Reparatursysteme einer Zelle erkannt werden, da dort keine komplementäre Basenpaarung vorliegt. Allerdings bliebe unklar, welche der beiden Möglichkeiten die richtige sei, wenn sich alter und neuer DNA-Strang nicht unterschieden. Da der alte Strang jedoch methyliert ist, der neue aber noch nicht, ist eine Unterscheidung möglich. Die DNA-Reparatursysteme von Bakterien können diesen hemimethylierten („halbmethylierten“) Zustand zur postreplikativen Fehlerkorrektur nutzen.
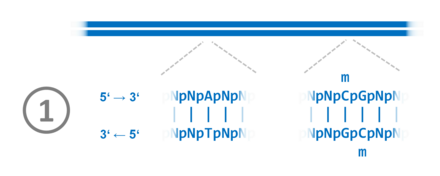
Bei Eukaryoten werden die Reparaturenzyme z. B. durch das Ringklemmenprotein (PCNA – Proliferating-Cell-Nuclear-Antigen) rekrutiert, welches die Stränge während der Replikation auseinander hält. Es gibt allerdings weitere Reparatur-Mechanismen. In den Zellen des Menschen wie auch anderer Säugetiere ist eine kurze Folge aus zwei Grundbausteinen (Nukleosiden) die Grundlage für eine DNA-Methylierung: das CpG-Dinukleotid (Desoxycytidin – Phosphorsäure – Desoxyguanosin). In der Aufeinanderfolge der beiden Nukleobasen Cytosin–Guanin wird das Cytosin methyliert. Bis auf einige Bereiche passiert dies fast in der gesamten menschlichen Erbsubstanz. Auch hier ist (wie bei den Bakterien) ein hemimethylierter Zustand für die postreplikative Reparatur entscheidend. Im Folgenden wird eine solche Reparatur anhand eines Beispiels vereinfacht, in sechs Schritten dargestellt:
 |
 |
|
1. Vor der DNA-Replikation sind im betrachteten Beispielabschnitt die CpG-Dinukleotide in beiden Strängen am Cytosin methyliert. 2. Während der DNA-Replikation kommt es zum Fehler: Statt eines Thymidintriphosphat-Moleküls wird ein Cytidintriphosphat verwendet. Dadurch wird an der entsprechenden Stelle die komplementäre Basenpaarung aufgehoben. 3. Nach Abschluss der DNA-Replikation im Beispielabschnitt liegt der DNA-Doppelstrang hemimethyliert vor. Das heißt, der alte Strang ist methyliert, der neue nicht. Ein Protein-Komplex bindet an die halbseitig methylierten CpG-Stellen (Hemi-mCpG-Np95-Dnmt1) und ermöglicht ein nachträgliche, neustrang-spezifische Reparatur bis in einen Bereich hinein, in welchem der DNA-Doppelstrang bereits beginnt, sich mit den Histonen zum Chromatin zu assemblieren.[1] 4. Die Stelle der Fehlpaarung wird erkannt. Von den beiden Möglichkeiten Adenosinmonophosphat und Cytidinmonophosphat wird das Cytidinmonophosphat ausgeschnitten, das im nichtmethylierten Strang liegt. Tymidintriphosphat wird verwendet, um die Lücke zu schließen. 5. Der reparierte DNA-Doppelstrang liegt im hemimethylierten Zustand vor 6. Durch die Übertragung von Methylgruppen auf die Nukleobase Cytosin in den CpG-Dinukleotiden wird der Grundzustand wiederhergestellt. |
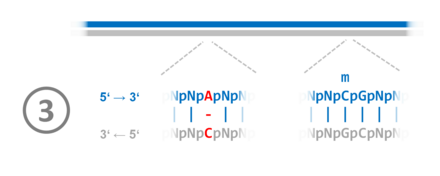 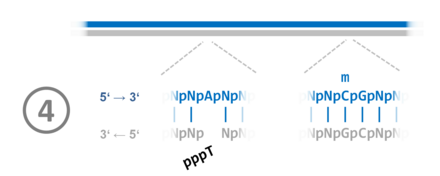 |
 |
 |
Bisherige Version[Bearbeiten | Quelltext bearbeiten]
Das ist die am 5. Nov. 2018 (06:30) ersetzte Version:
- (Aktuell | Vorherige) 17:56, 4. Nov. 2018 Dirk123456 (Diskussion | Beiträge) . . (79.853 Bytes) (-1) . . (→DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese: verrutschte Methylgruppen in der Textabbildung, Schritt 4) (rückgängig) [automatisch gesichtet]
DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese[Bearbeiten | Quelltext bearbeiten]
Die identische Verdopplung der Desoxyribonukleinsäure (DNA-Replikation) ist eine wesentliche Voraussetzung für die Zellteilung und damit für die Vermehrung. Die DNA-Replikation wird dadurch gewährleistet, dass Enzyme (die DNA-abhängigen DNA-Polymerasen) den bereits vorhandenen alten Strang „lesen“ und dabei den neuen Strang „schreiben“. Dabei können Fehler auftreten. Die Fehlerstellen können durch die DNA-Reparatursysteme einer Zelle erkannt werden, da dort keine komplementäre Basenpaarung vorliegt. Allerdings bliebe unklar, welche der beiden Möglichkeiten die richtige sei, wenn sich alter und neuer DNA-Strang nicht unterschieden. Da der alte Strang jedoch methyliert ist, der neue aber noch nicht, ist eine Unterscheidung möglich. Die DNA-Reparatursysteme von Bakterien können diesen hemimethylierten („halbmethylierten“) Zustand zur postreplikativen Fehlerkorrektur nutzen.
Bei Eukaryoten werden die Reparaturenzyme z. B. durch das Ringklemmenprotein (PCNA – Proliferating-Cell-Nuclear-Antigen) rekrutiert, welches die Stränge während der Replikation auseinander hält. Es gibt allerdings weitere Reparatur-Mechanismen. In den Zellen des Menschen wie auch anderer Säugetiere ist eine kurze Folge aus zwei Grundbausteinen (Nukleosiden) die Grundlage für eine DNA-Methylierung: das CpG-Dinukleotid (Desoxycytidin – Phosphorsäure – Desoxyguanosin). In der Aufeinanderfolge der beiden Nukleobasen Cytosin–Guanin wird das Cytosin methyliert. Bis auf einige Bereiche passiert dies fast in der gesamten menschlichen Erbsubstanz. Auch hier ist (wie bei den Bakterien) ein hemimethylierter Zustand für die postreplikative Reparatur entscheidend:
1. Vor der DNA-Replikation sind im betrachteten Beispielabschnitt die CpG-Dinukleotide in beiden Strängen am Cytosin methyliert:
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | | | | | | | |
... TpTpGpApGpCpApCpGpTpTpTpGpCpCpApAp...
m m
2. Während der DNA-Replikation kommt es zum Fehler: Statt eines Thymidintriphosphat-Moleküls wird ein Cytidintriphosphat verwendet. Dadurch wird an der entsprechenden Stelle die komplementäre Basenpaarung aufgehoben:
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | |
CpApCpGpCpTpTpGpCpCpApAp...
3. Nach Abschluss der DNA-Replikation im Beispielabschnitt liegt der DNA-Doppelstrang hemimethyliert vor. Das heißt, der alte Strang ist methyliert, der neue nicht. Ein Protein-Komplex bindet an die halbseitig methylierten CpG-Stellen (Hemi-mCpG-Np95-Dnmt1) und ermöglicht ein nachträgliche, neustrang-spezifische Reparatur bis in einen Bereich hinein, in welchem der DNA-Doppelstrang bereits beginnt, sich mit den Histonen zum Chromatin zu assemblieren.[1]
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | | | | | | |
... TpTpGpApGpCpApCpGpCpTpTpGpCpCpApAp...
4. Die Stelle der Fehlpaarung wird erkannt. Von den beiden Möglichkeiten Adenosinmonophosphat und Cytidinmonophosphat wird das Cytidinmonophosphat ausgeschnitten, das im nichtmethylierten Strang liegt. Tymidintriphosphat wird verwendet, um die Lücke zu schließen:
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | | | | | | |
... TpTpGpApGpCpApCpGp TpTpGpCpCpApAp...
pppT
5. Der reparierte DNA-Doppelstrang liegt im hemimethylierten Zustand vor:
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | | | | | | | |
... TpTpGpApGpCpApCpGpTpTpTpGpCpCpApAp...
6. Durch die Übertragung von Methylgruppen auf die Nukleobase Cytosin in den CpG-Dinukleotiden wird der Grundzustand wiederhergestellt:
m m
...pApApCpTpCpGpTpGpCpApApApCpGpGpTpT ...
| | | | | | | | | | | | | | | | |
... TpTpGpApGpCpApCpGpTpTpTpGpCpCpApAp...
m m
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c Keh-Yang Wang, Chun-Chang Chen, Shih-Feng Tsai, Che-Kun James Shen: Epigenetic Enhancement of the Post-replicative DNA Mismatch Repair of Mammalian Genomes by a Hemi-mCpG-Np95-Dnmt1 Axis. In: Scientific Reports. 6, 2016, S. 37490, doi:10.1038/srep37490. PMC 5122852 (freier Volltext). PMID 27886214.
- ↑ Y. Iwasaki, T. Abe, N. Okada, K. Wada, Y. Wada, T. Ikemura: Evolutionary changes in vertebrate genome signatures with special focus on coelacanth. In: DNA research : an international journal for rapid publication of reports on genes and genomes. Band 21, Nummer 5, Oktober 2014, S. 459–467, doi:10.1093/dnares/dsu012, PMID 24800745, PMC 4195492 (freier Volltext).
- ↑ X. He, D. Tillo, J. Vierstra, K. S. Syed, C. Deng, G. J. Ray, J. Stamatoyannopoulos, P. C. FitzGerald, C. Vinson: Methylated Cytosines Mutate to Transcription Factor Binding Sites that Drive Tetrapod Evolution. In: Genome biology and evolution. Band 7, Nummer 11, Oktober 2015, S. 3155–3169, doi:10.1093/gbe/evv205, PMID 26507798, PMC 4994754 (freier Volltext).
- ↑ S. Hu, J. Wan, Y. Su, Q. Song, Y. Zeng, H. N. Nguyen, J. Shin, E. Cox, H. S. Rho, C. Woodard, S. Xia, S. Liu, H. Lyu, G. L. Ming, H. Wade, H. Song, J. Qian, H. Zhu: DNA methylation presents distinct binding sites for human transcription factors. In: eLife. Band 2, September 2013, S. e00726, doi:10.7554/eLife.00726, PMID 24015356, PMC 3762332 (freier Volltext).