DNA-bindende Proteine
Ein DNA-bindendes Protein ist ein Protein, das an DNA bindet.[1][2][3]
Eigenschaften[Bearbeiten | Quelltext bearbeiten]
DNA-bindende Proteine kommen in allen Lebewesen und DNA-Viren vor. Sie besitzen mindestens eine Proteindomäne, welche an DNA binden kann. Die Bindung kann dabei an verschiedenen funktionellen Gruppen erfolgen. Das Rückgrat der DNA besteht aus sich abwechselnden Phosphat- und Desoxyribose-Einheiten. Durch die Phosphatgruppen ist die DNA proportional zur Kettenlänge mit negativen Ladungen versehen, an die unter anderem Proteindomänen mit positiv-geladenen Aminosäuren (z. B. Lysin, Arginin) oder Proteindomänen mit negativ geladenen Aminosäuren mit komplexierten Kationen binden können (z. B. Mg2+- oder Zn2+-Komplexe).[4] Da das Desoxyribosephosphat-Rückgrat sich ständig wiederholt, kann ein ausschließlich an das Rückgrat bindendes Protein nicht an eine bestimmte DNA-Sequenz binden (keine Sequenzspezifität). Eine sequenzspezifische Bindung erfolgt durch zumindest teilweise Bindung an eine bestimmte Folge von Nukleinbasen, teilweise kann auch das Rückgrat gebunden werden.
Sequenzspezifität[Bearbeiten | Quelltext bearbeiten]
DNA-bindende Proteine ohne Sequenzspezifität sind z. B. die Polymerasen, Helicasen und generell Proteine, die an der DNA entlanggleiten (z. B. mit DNA-Klammer). Sequenzspezifisch DNA-bindende Proteine sind z. B. Transkriptionsfaktoren, die an definierten Stellen (Promotor) eine Genexpression auslösen.[5] Aufgrund des höheren Anteils an Nukleinbasen in der Oberfläche binden sequenzspezifische DNA-bindende Moleküle eher in der großen Furche der DNA-Doppelhelix.[6] Während manche sequenzspezifisch DNA-bindende Proteine bevorzugt einzelsträngige DNA binden (z. B. Einzelstrang-bindendes Protein), binden andere doppelsträngige DNA (die meisten) und einige wenige auch Heteroduplexe aus DNA und RNA (z. B. Telomerase, Reverse Transkriptasen, RNase H).
Bindung einzelsträngiger DNA[Bearbeiten | Quelltext bearbeiten]
Einzelsträngige DNA kommt in Eukaryoten dauerhaft an den Telomeren vor und vorübergehend unter anderem bei der Replikation, der Transkription, der Rekombination und der DNA-Reparatur vor,[7] z. B. das Einzelstrang-bindende Protein und manche DNA-Reparaturenzyme.[8][9]
Bindung doppelsträngiger DNA[Bearbeiten | Quelltext bearbeiten]
Doppelsträngige DNA mit komplementärer Basenpaarung bildet eine Doppelhelix aus (B-DNA). Diese DNA-Doppelhelix besitzt eine große und eine kleine Furche. Die kleine Furche besitzt einen kleineren Anteil der Nukleinbasen in der Oberfläche des Moleküls, weshalb sie sich weniger für eine Sequenz-spezifische Bindung eignet. Verschiedene DNA-bindende Moleküle wie Lexitropsine, Netropsin,[10] Distamycin, Hoechst 33342, Pentamidin, DAPI oder SYBR Green I binden sequenzunabhängig an die kleine Furche doppelsträngiger DNA.[11] Doppelstrang-bindende Proteine sind unter anderem Histone[12] bzw. DNA-bindendes Protein H-NS,[13] High-Mobility-Group-Proteine,[14][15] DNA-Polymerasen, DNA-abhängige RNA-Polymerasen, Helicasen, Topoisomerasen, Gyrasen, Ligasen, Polynukleotid-Kinasen, Nukleasen, manche DNA-Reparaturenzyme. Sequenzspezifisch dsDNA-bindende Proteine sind unter anderem Transkriptionsfaktoren und manche Endonukleasen.[16][17] Prokaryotische Transkriptionsfaktoren sind meistens kleiner als die von ihnen kontrollierten Genexpressionsprodukte, während eukaryotische Transkriptionsfaktoren meistens größer als deren kontrollierten Produkte sind und gelegentlich mehrere Kopien einer DNA-bindenden Domäne besitzen.[18]
DNA-bindende Proteindomänen[Bearbeiten | Quelltext bearbeiten]
Typische Proteindomänen bei dsDNA-bindenden Proteinen sind die Zinkfingerdomäne, die AT-Haken, die DNA-Klammer und das Helix-Turn-Helix-Motiv zur DNA-Bindung oder die Leucinzipperdomäne (bZIP) zur Dimerisierung. Bei einzelsträngiger DNA wurde unter anderem die OB-Faltungsdomäne beschrieben.[19]
Identifikation[Bearbeiten | Quelltext bearbeiten]
Methoden zur Bestimmung von Protein-DNA-Wechselwirkungen (gebundene DNA-Sequenz, DNA-bindende Proteine) sind z. B. EMSA, DNase Footprinting Assay, Filterbindungstest, DPI-ELISA, DamID, SELEX, ChIP, ChIP-on-Chip oder ChIP-Seq und DAP-Seq.[20][21]
Modellierung[Bearbeiten | Quelltext bearbeiten]
Verschiedene Ansätze zur molekularen Modellierung wurden beschrieben.[22][23] Ebenso wurden Algorithmen zur Bestimmung der gebundenen DNA-Sequenz entwickelt.[24]
Modifikation[Bearbeiten | Quelltext bearbeiten]
Im Zuge eines Proteindesigns können z. B. Zinkfingerproteine oder TALENs entworfen werden. Mit der CRISPR/Cas-Methode können anhand einer komplementären RNA-Sequenz entsprechende DNA-Sequenzen gebunden werden.
Formen[Bearbeiten | Quelltext bearbeiten]
-
Cro im Komplex mit DNA
-
DNA (orange) mit Histonen (blau)
-
DNA-Klammer
Weblinks[Bearbeiten | Quelltext bearbeiten]
- Abalone tool for modeling DNA-ligand interactions.
- DBD database of predicted transcription factors Uses a curated set of DNA-binding domains to predict transcription factors in all completely sequenced genomes
- MeSH DNA-bindende Proteine
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ A. A. Travers: DNA-protein interactions. Springer, London 1993, ISBN 0-412-25990-7.
- ↑ C. O. Pabo, R. T. Sauer: Protein-DNA recognition. In: Annu. Rev. Biochem. Band 53, Nr. 1, 1984, S. 293–321, doi:10.1146/annurev.bi.53.070184.001453, PMID 6236744.
- ↑ R. E. Dickerson: The DNA helix and how it is read. In: Sci Am. Band 249, Nr. 6, 1983, S. 94–111, doi:10.1038/scientificamerican1283-94.
- ↑ K. Luger, A. Mäder, R. Richmond, D. Sargent, T. Richmond: Crystal structure of the nucleosome core particle at 2.8 A resolution. In: Nature. Band 389, Nr. 6648, 1997, S. 251–260, doi:10.1038/38444, PMID 9305837.
- ↑ Z. Li, S. Van Calcar, C. Qu, W. Cavenee, M. Zhang, B. Ren: A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells. In: Proc Natl Acad Sci USA. Band 100, Nr. 14, 2003, S. 8164–8169, doi:10.1073/pnas.1332764100, PMID 12808131, PMC 166200 (freier Volltext).
- ↑ C. Pabo, R. Sauer: Protein-DNA recognition. In: Annu Rev Biochem. Band 53, Nr. 1, 1984, S. 293–321, doi:10.1146/annurev.bi.53.070184.001453, PMID 6236744.
- ↑ T. H. Dickey, S. E. Altschuler, D. S. Wuttke: Single-stranded DNA-binding proteins: multiple domains for multiple functions. In: Structure (London, England : 1993). Band 21, Nummer 7, Juli 2013, S. 1074–1084, doi:10.1016/j.str.2013.05.013. PMID 23823326. PMC 3816740 (freier Volltext).
- ↑ C. Iftode, Y. Daniely, J. Borowiec: Replication protein A (RPA): the eukaryotic SSB. In: Crit Rev Biochem Mol Biol. Band 34, Nr. 3, 1999, S. 141–180, doi:10.1080/10409239991209255, PMID 10473346.
- ↑ J. Kur, M. Olszewski, A. Dugoecka, P. Filipkowski: Single-stranded DNA-binding proteins (SSBs) – sources and applications in molecular biology. In: Acta biochimica Polonica. Band 52, Nummer 3, 2005, S. 569–574. PMID 16082412.
- ↑ C. Zimmer, U. Wähnert: Nonintercalating DNA-binding ligands: specificity of the interaction and their use as tools in biophysical, biochemical and biological investigations of the genetic material. In: Prog. Biophys. Mol. Biol. Band 47, Nr. 1, 1986, S. 31–112, doi:10.1016/0079-6107(86)90005-2, PMID 2422697.
- ↑ P. B. Dervan: Design of sequence-specific DNA-binding molecules. In: Science. Band 232, Nr. 4749, April 1986, S. 464–471, doi:10.1126/science.2421408, PMID 2421408.
- ↑ K. Sandman, S. Pereira, J. Reeve: Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome. In: Cell Mol Life Sci. Band 54, Nr. 12, 1998, S. 1350–1364, doi:10.1007/s000180050259, PMID 9893710.
- ↑ R. T. Dame: The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin. In: Mol. Microbiol. Band 56, Nr. 4, 2005, S. 858–870, doi:10.1111/j.1365-2958.2005.04598.x, PMID 15853876.
- ↑ J. Thomas: HMG1 and 2: architectural DNA-binding proteins. In: Biochem Soc Trans. Band 29, Pt 4, 2001, S. 395–401, doi:10.1042/BST0290395, PMID 11497996.
- ↑ R. Grosschedl, K. Giese, J. Pagel: HMG domain proteins: architectural elements in the assembly of nucleoprotein structures. In: Trends Genet. Band 10, Nr. 3, 1994, S. 94–100, doi:10.1016/0168-9525(94)90232-1, PMID 8178371.
- ↑ L. Myers, R. Kornberg: Mediator of transcriptional regulation. In: Annu Rev Biochem. Band 69, Nr. 1, 2000, S. 729–749, doi:10.1146/annurev.biochem.69.1.729, PMID 10966474.
- ↑ B. Spiegelman, R. Heinrich: Biological control throughs regulated transcriptional coactivators. In: Cell. Band 119, Nr. 2, 2004, S. 157–167, doi:10.1016/j.cell.2004.09.037, PMID 15479634.
- ↑ V. Charoensawan, D. Wilson, S. A. Teichmann: Genomic repertoires of DNA-binding transcription factors across the tree of life. In: Nucleic acids research. Band 38, Nummer 21, November 2010, S. 7364–7377, doi:10.1093/nar/gkq617. PMID 20675356. PMC 2995046 (freier Volltext).
- ↑ N. W. Ashton, E. Bolderson, L. Cubeddu, K. J. O’Byrne, D. J. Richard: Human single-stranded DNA binding proteins are essential for maintaining genomic stability. In: BMC molecular biology. Band 14, 2013, S. 9, doi:10.1186/1471-2199-14-9. PMID 23548139. PMC 3626794 (freier Volltext).
- ↑ H. L. Brand, S. B. Satbhai, H. Ü. Kolukisaoglu, D. Wanke: Limits And Prospects Of Methods For The Analysis Of DNA-Protein Interaction. Bentham eBook, S. 124–148, abgerufen am 21. Oktober 2020 (englisch).
- ↑ M. F. Carey, C. L. Peterson, S. T. Smale: Experimental strategies for the identification of DNA-binding proteins. In: Cold Spring Harbor protocols. Band 2012, Nummer 1, Januar 2012, S. 18–33, doi:10.1101/pdb.top067470. PMID 22194258.
- ↑ V. B. Teif, K. Rippe: Statistical-mechanical lattice models for protein-DNA binding in chromatin. In: Journal of Physics: Condensed Matter. 2010, arxiv:1004.5514.
- ↑ K. C. Wong, T. M. Chan, C. Peng, Y. Li, Z. Zhang: DNA Motif Elucidation using belief propagation. In: Nucleic Acids Research. Advanced Online June 2013; doi:10.1093/nar/gkt574. PMID 23814189
- ↑ G. D. Stormo: DNA binding sites: representation and discovery. In: Bioinformatics. Band 16, Nummer 1, Januar 2000, S. 16–23. PMID 10812473.
- ↑ Created from PDB 1LMB
- ↑ Created from PDB 1RVA
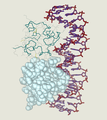

![Der Lambda-Repressor mit Helix-Turn-Helix-Motiv an DNA gebunden[25]](http://upload.wikimedia.org/wikipedia/commons/thumb/8/8f/Lambda_repressor_1LMB.png/82px-Lambda_repressor_1LMB.png)
![Das Restriktionsenzym EcoRV (grün) mit DNA.[26]](http://upload.wikimedia.org/wikipedia/commons/thumb/d/dc/EcoRV_1RVA.png/120px-EcoRV_1RVA.png)
