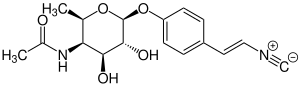
Rhabduscin
| Strukturformel | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||
| Allgemeines | ||||||||||
| Name | Rhabduscin | |||||||||
| Summenformel | C17H20N2O5 | |||||||||
| Externe Identifikatoren/Datenbanken | ||||||||||
| ||||||||||
| Eigenschaften | ||||||||||
| Molare Masse | 332,40 g·mol−1 | |||||||||
| Sicherheitshinweise | ||||||||||
| ||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | ||||||||||
Rhabduscin ist ein bakterielles Glycosid, dessen Aglycon über eine Isonitril-Funktion verfügt. Dasselbe Aglycon kommt auch noch in Byelyankacin vor.[2] Das Glykon (die Zuckerkomponente) ist 4-Acetamido-4-desoxy-α-D-fucose, ein Derivat der Fucose, bei der eine Hydroxygruppe durch eine Acetamid-Gruppe substituiert ist.
Vorkommen[Bearbeiten | Quelltext bearbeiten]
Rhabduscin kommt in den meisten Bakterien der Gattungen Xenorhabdus und Photorhabdus vor[3], darunter Xenorhabdus nematophila und Xenorhabdus poinarii[4], sowie in Photorhabdus luminescens[5], Photorhabdus asymbiotica und Photorhabdus temperata.[6] Dabei liegt es vor allem an die Außenseite bakterieller Zellen gebunden vor.[7]
Xenorhabdus sind für Insekten pathogen. Teil der Immunantwort von Insekten ist das sogenannte Phenoloxidase-System bei dem durch eine Phenoloxidase phenolische Verbindungen aus Pathogenen zu Melaninen polymerisiert werden, wodurch Wunden verschlossen und verbleibende Pathogene eingeschlossen werden. Rhabduscin hemmt neben weiteren Verbindungen (z. B. auch Benzaldehyd und Terephthalsäure) die Phenoloxidase, wodurch diese Bakterien die Immunantwort zum Teil umgehen können.[4] Rhabduscin ist dabei ein wichtiger Virulenzfaktor.[7]
Biosynthese[Bearbeiten | Quelltext bearbeiten]
In Xenorhabdus nematophila wurde ein Gen-Cluster identifiziert, der für die Biosynthese von Rhabduscin verantwortlich ist. Er enthält zwei Gene (isnA und isnB), die für Proteine codieren, welche für die Synthese des Aglycons mit der Isonotril-Funktion nötig sind. Ein drittes Gen codiert für eine Glycosyltransferase. Durch heterologe Expression dieser Gene konnte Escherichia coli Rhabduscin bilden.[4][5] Homologe der Gene isnA und isnB wurden auch in vielen anderen Bakterien entdeckt, darunter auch solche die für Menschen pathogen sind, darunter Pseudomonas aeruginosa.[4]
Die Biosynthese geht vermutlich von Tyrosin aus, bei dem zunächst die Aminogruppe in eine Isonitril-Gruppe umgewandelt wird. Das Stickstoffatom stammt also vom Tyrosin.[3][8] Durch Decarboxylierung und Bildung einer Doppelbindung entsteht dann das Aglycon mit der Vinyl-Isonitril-Substruktur. Zuletzt wird dieses glycosyliert. Für den zweiten Schritt ist ein Non-Häm-Eisen-Protein verantwortlich.[8]

Die entsprechenden Gene in Photorhabdus luminescens liegen nicht gemeinsam an einem Ort im Genom vor, vielmehr treten dort nur isnA und isnB gemeinsam auf und getrennt davon zwei Glycosyltransferasen, die Rhabduscin und Byelyankacin bilden.[7]
Durch Prolin, welches in der Hämolymphe von Insekten in großer Menge vorkommt, wird sowohl bei Xenorhabdus nematophila als auch bei Photorhabdus luminescens die Biosynthese von Rhabduscin hochreguliert.[5][7]
Eigenschaften[Bearbeiten | Quelltext bearbeiten]
Rhabduscin ist ein starker Inhibitor (IC50 im nanomolaren Bereich) sowohl der Phenoloxidase von Insekten als auch der Tyrosinase von Pilzen. Es wurde gezeigt, dass die Isonitril-Funktion für eine starke Hemmung der Phenoloxidase unbedingt nötig ist (Analoga mit einer Nitril- oder Azid-Funktion haben eine deutlich schwächere Wirkung), was darauf hindeutet, dass Rhabduscin über die Isonitril-Gruppe an Kupfer bindet, das im Enzym enthalten ist.[7]
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ Dieser Stoff wurde in Bezug auf seine Gefährlichkeit entweder noch nicht eingestuft oder eine verlässliche und zitierfähige Quelle hierzu wurde noch nicht gefunden.
- ↑ Jason M. Crawford, Jon Clardy: Bacterial symbionts and natural products. In: Chemical Communications. Band 47, Nr. 27, 2011, S. 7559, doi:10.1039/c1cc11574j, PMID 21594283, PMC 3174269 (freier Volltext).
- ↑ a b Yi-Ming Shi, Helge B. Bode: Chemical language and warfare of bacterial natural products in bacteria–nematode–insect interactions. In: Natural Product Reports. Band 35, Nr. 4, 2018, S. 309–335, doi:10.1039/C7NP00054E.
- ↑ a b c d Maria Eugenia Nuñez-Valdez, Anne Lanois, Sylvie Pagès, Bernard Duvic, Sophie Gaudriault: Inhibition of Spodoptera frugiperda phenoloxidase activity by the products of the Xenorhabdus rhabduscin gene cluster. In: PLOS ONE. Band 14, Nr. 2, 22. Februar 2019, S. e0212809, doi:10.1371/journal.pone.0212809, PMID 30794697, PMC 6386379 (freier Volltext).
- ↑ a b c Jason M. Crawford, Renee Kontnik, Jon Clardy: Regulating Alternative Lifestyles in Entomopathogenic Bacteria. In: Current Biology. Band 20, Nr. 1, Januar 2010, S. 69–74, doi:10.1016/j.cub.2009.10.059, PMID 20022247, PMC 2821981 (freier Volltext).
- ↑ The Molecular Biology of Photorhabdus Bacteria. In: Current Topics in Microbiology and Immunology. Band 402. Springer International Publishing, Cham 2017, ISBN 978-3-319-52714-7, doi:10.1007/978-3-319-52715-4.
- ↑ a b c d e Jason M. Crawford, Cyril Portmann, Xu Zhang, Maarten B. J. Roeffaers, Jon Clardy: Small molecule perimeter defense in entomopathogenic bacteria. In: Proceedings of the National Academy of Sciences. Band 109, Nr. 27, 3. Juli 2012, doi:10.1073/pnas.1201160109, PMID 22711807, PMC 3390839 (freier Volltext).
- ↑ a b Cheng-Ping Yu, Yijie Tang, Lide Cha, Sergey Milikisiyants, Tatyana I. Smirnova, Alex I. Smirnov, Yisong Guo, Wei-chen Chang: Elucidating the Reaction Pathway of Decarboxylation-Assisted Olefination Catalyzed by a Mononuclear Non-Heme Iron Enzyme. In: Journal of the American Chemical Society. Band 140, Nr. 45, 14. November 2018, S. 15190–15193, doi:10.1021/jacs.8b10077, PMID 30376630, PMC 6480304 (freier Volltext).