Sporosarcina ureae
| Sporosarcina ureae | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Sporosarcina ureae | ||||||||||||
| (Beijerinck 1901) Kluyver und van Niel 1936 |
Sporosarcina ureae ist eine Bakterienart. Sie ist die Typusart ihrer Gattung und ist mit Bacillus-Arten verwandt. Wie diese kommt sie im Boden vor. Außerdem ist sie in der Lage, Harnstoff abzubauen, diese Besonderheit ist im Artnamen verzeichnet (lateinisch urea, „Harnstoff“). Der Gattungsname leitet sich vom griechischen Wort spora („Spore“) und dem lateinischen Wort sarcina („Paket“, „Bündel“) ab und bezieht sich darauf, dass sie Endosporen bildet und auf die typische Anordnung der Zellen.
Merkmale[Bearbeiten | Quelltext bearbeiten]
Erscheinungsbild[Bearbeiten | Quelltext bearbeiten]
Die Zellen von Sporosarcina ureae sind grampositiv und kokkenförmig. Die Zellteilung erfolgt in zwei oder drei aufeinander stehenden Ebenen, so werden Tetraden oder auch Pakete von acht oder mehr Zellen gebildet.[1] Der Durchmesser der Zellen liegt zwischen 1,0 und 2,5 μm. S. ureae ist durch einige, zufällig positionierte, Flagellen motil, sie kann sich selbst bewegen. Wie alle Arten der Gattung bildet S. ureae Endosporen als Überdauerungsform. Der Durchmesser der Endosporen liegt zwischen 0,5 und 1,5 μm.[2] Die Endosporen sind kugelförmig und werden zentral oder lateral in der Mutterzelle gebildet. Im lichtmikroskopischen Bild sind sie als helle, lichtbrechende Formen zu erkennen.[3]
Auf festen Nährböden wachsen die Zellen zu grauen Kolonien heran. Diese sind opak, in der Aufsicht rund und mit einer klaren Begrenzung, von der Seite betrachtet leicht konvex. Auf einigen Nährböden werden gelbliche, bräunliche oder orange Pigmente gebildet.[3]
Wachstum und Stoffwechsel[Bearbeiten | Quelltext bearbeiten]
Die Art Sporosarcina ureae ist heterotroph, sie führt keine Photosynthese durch. Der Stoffwechsel beruht auf der Atmung. Die Art ist strikt aerob, d. h. auf Sauerstoff angewiesen, Wachstum unter Sauerstoffausschluss findet nicht statt. Der Katalase- und der Oxidase-Test fällt positiv aus.[3] Der pH-Wert für bestes Wachstum ist 7. Die Art ist alkalitolerant und wächst auch bei pH-Werten von 9 bis 10.[4] Sie ist mesophil, die optimale Temperatur für das Wachstum liegt bei 25 °C. Bei 37 °C kann noch schwaches Wachstum erfolgen, bei höheren Temperaturen nicht mehr. S. ureae lässt sich in einem Nährmedium kultivieren, das neben Pepton und Hefeextrakt noch Harnstoff oder Ammoniumchlorid enthält.[3]
Die Sporenbildung erfolgt nicht automatisch, sondern wird durch bestimmte Umwelteinflüsse auf die Zellen ausgelöst. Die Bildung von Endosporen erfolgt beispielsweise, wenn die Temperatur für die Inkubation unter 22 °C liegt. Die Endosporen sind hitzeresistent, sie überstehen eine Erhitzung auf 80 °C für 10 Minuten und können danach noch auskeimen. Wie bei bakteriellen Endosporen üblich, enthalten sie Dipicolinsäure.[3]
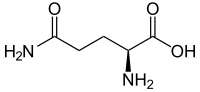
Wie im Epitheton vermerkt, kann S. ureae Harnstoff (lateinisch urea) mit Hilfe des Enzyms Urease abbauen. Dabei wird der Harnstoff (NH2-CO-NH2) unter Reaktion mit Wasser (H2O) „gespalten“ (hydrolysiert), es entstehen – je nach pH-Wert der Lösung – Ammoniak (NH3) oder Ammonium-Ionen (NH4+) und Kohlenstoffdioxid (CO2) bzw. Carbonat- oder Hydrogencarbonat-Ionen:[1]
bzw.
S. ureae kann auch direkt Ammonium-Ionen aus dem Nährmedium verwenden, wenn dieses z. B. Ammoniumchlorid (NH4Cl) enthält. Im Stoffwechsel wird Ammonium assimiliert, um daraus Glutaminsäure und Glutamin, zwei Aminosäuren herzustellen. Biochemische und stoffwechselphysiologische Untersuchungen zeigen, dass S. ureae dabei einen Stoffwechselweg ähnlich dem GS-GOGAT-Weg durchführt, benannt nach den beteiligten Enzymen Glutamin-Synthetase und Glutamin-Oxoglutarat-Aminotransferase. Allerdings ist keine Aktivität der Glutamin-Synthetase (aktuelle Bezeichnung: Glutamat-Ammonium-Ligase) erkennbar, jedoch eine GOGAT-Aktivität, hierdurch wird von der Aminosäure Glutamin eine Aminogruppe auf das 2-Oxoglutarat (α-Ketoglutarat, das Anion der 2-Oxoglutarsäure) übertragen, wobei Glutamat (das Anion der Glutaminsäure) entsteht. Genauso wird die Rückreaktion katalysiert:[5]
- 2 Glutamat → Glutamin + 2-Oxoglutarat + e−
- Vereinfachte Reaktionsgleichung, das beteiligte Enzym ist die Glutamin-Oxoglutarat-Aminotransferase, die Elektronen (e−) werden auf den Elektronen-Akzeptor NAD+ (oxidierte Form des Nicotinamidadenindinukleotids) übertragen.
Außerdem lässt sich Glutamatdehydrogenase (GDH) nachweisen, dieses Enzym katalysiert die Umwandlung von Glutamat in 2-Oxoglutarat und Ammonium. Da die GDH auch die Rückreaktion katalysiert, ist dieses Enzym primär an der Assimilation von Ammonium beteiligt:[5]
- 2-Oxoglutarat + NH4+ + e− → Glutamat + H2O
- Vereinfachte Reaktionsgleichung, das beteiligte Enzym ist die Glutamatdehydrogenase, die Elektronen (e−) werden von dem Elektronen-Donator NADH (reduzierte Form des Nicotinamidadenindinukleotids) übertragen.
Genetik[Bearbeiten | Quelltext bearbeiten]
Das Genom des Stammes Sporosarcina ureae DSM 2281 wurde im Jahr 2013 vollständig sequenziert,[6] dabei handelt es sich um den Typusstamm der Art.[7] Das Genom weist eine Größe von 3319 Kilobasenpaaren (kb) auf,[6] das ist etwa 70 % der Genomgröße von Escherichia coli. Das Genom wurde im Rahmen des KMG-Projektes erforscht, bei dem 1000 Genome von Mikroorganismen (one thousand microbial genomes) sequenziert werden, um eine „Genom-Enzyklopädie“ von Typusstämmen (Genomic Encyclopedia of Type Strains) zu erstellen. Auf diese Weise sollen durch phylogenetische Untersuchungen die Verwandtschaftsbeziehungen der Mikroorganismen geklärt werden.[8] Das Ergebnis der Sequenzierung zeigt einen niedrigen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA, er liegt bei etwa 41 Mol-Prozent.[6] Ein niedriger GC-Gehalt ist typisch für die Vertreter der Ordnung Bacillales und anderer Firmicutes.[1]
Pathogenität[Bearbeiten | Quelltext bearbeiten]
Sporosarcina ureae ist nicht pathogen („krankheitserregend“), sie wird durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 1 zugeordnet.[9]
Nachweise[Bearbeiten | Quelltext bearbeiten]
Biochemische Merkmale, wie beispielsweise die vorhandenen Enzyme können in einer „Bunten Reihe“ zur Identifizierung von Sporosarcina ureae verwendet werden. Neben dem positiven Katalase- und Oxidase-Test können folgende Merkmale zur Unterscheidung zu den anderen Arten herangezogen werden: Sie kann Nitrat zu Nitrit reduzieren, bei dieser Denitrifikation wird jedoch kein Gas (molekularer Stickstoff) gebildet. Sie besitzt das Enzym Urease. Sie kann nicht Stärke, Gelatine oder Casein durch Hydrolyse verwerten. Die Voges-Proskauer-Reaktion und der Indol-Test verlaufen negativ.[3]
Die Zellwand von S. ureae besteht aus dem für grampositive Bakterien typischen Peptidoglycan. In den darin enthaltenen Peptiden ist die Kombination der Aminosäuren Lysin, Glycin und D-Glutaminsäure typisch und ermöglicht die Unterscheidung von anderen Sporosarcina-Arten. Die Lipide in der Zellmembran enthalten Phosphatidylethanolamine. Die hauptsächlich vorkommenden Menachinone sind vom Typ MK-7.[3]
Ein Selektivmedium zur Isolierung ist nicht vorhanden, jedoch kann man sich die Urease-Aktivität von S. ureae zu Nutze machen. Wenn das Nährmedium einen relativ hohen Gehalt an Harnstoff (3–5 %) aufweist, kann sie darauf wachsen und den Harnstoff verwerten. Die meisten ureasepositive Bakterien werden durch einen hohen Harnstoffgehalt im Wachstum gehemmt, dies trifft u. a. auf Bacillus mycoides zu, der häufig neben S. ureae in Bodenproben vorhanden ist.[3]
Vorkommen und Ökologie[Bearbeiten | Quelltext bearbeiten]
Sporosarcina ureae gehört zu den Bakterien, die Harnstoff verwerten können, sie verfügt über das Enzym Urease. Sie kommt häufig im Erdboden vor. Sie bildet höchste Populationsdichten in Böden, die Urineinfluss unterliegen. Hierzu zählen z. B. Viehweiden,[1] sowie das Erdreich von Bäumen, an denen Hunde urinieren.[3] Da Harnstoff bei vielen Lebewesen das Endprodukt des Stoffwechsels von Stickstoffverbindungen ist, ist er ein typischer Bestandteil des Urins. S. ureae spielt für den Abbau von Urin in der Umwelt daher eine wichtige Rolle.[1] Sie wird auch in Jauche gefunden.[4]
Systematik[Bearbeiten | Quelltext bearbeiten]
Die Erstbeschreibung erfolgte 1901 durch Martinus Willem Beijerinck, der das Bakterium als Planosarcina ureae benannte. Wegen der typischen Anordnung der Zellen, die als Sarcinen bezeichnet werden, wurde das Bakterium 1911 als Sarcina ureae der Gattung Sarcina zugeordnet. Erst durch die Beschreibung einer eigenständigen Gattung Sporosarcina (1936) erfolgte die aktuell immer noch gültige Zuordnung zur Gattung.[3][10] In den 1970er Jahren wurde erkannt, dass S. ureae mit Bacillus-Arten, besonders mit Bacillus pasteurii verwandt ist. Wie diese ist sie grampositiv, wächst strikt aerob, ist durch Geißeln beweglich und bildet Endosporen aus. Einen Unterschied gibt es in der Zellform, bei Bacillus-Arten handelt es sich um stäbchenförmige Bakterien.[3] Die Verwandtschaft wurde 1992 durch genetische Untersuchungen bestätigt, dabei wurde die 16S rRNA, ein für Prokaryoten typischer Vertreter der ribosomalen RNA untersucht.[11] In der Systematik der Bakterien wurde dies durch Zuordnung zu der gemeinsamen Ordnung Bacillales in der Abteilung der Firmicutes berücksichtigt.[7] Die Art Sporosarcina ureae zählt zu der Familie der Planococcaceae.[2][7]
Quellen[Bearbeiten | Quelltext bearbeiten]
Literatur[Bearbeiten | Quelltext bearbeiten]
- Paul Vos, George Garrity, Dorothy Jones, Noel R. Krieg, Wolfgang Ludwig, Fred A. Rainey, Karl-Heinz Schleifer, William B. Whitman: Bergey’s Manual of Systematic Bacteriology: Volume 3: The Firmicutes. Springer, 2009, ISBN 978-0-387-95041-9.
- Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 4: Bacteria: Firmicutes, Cyanobacteria. 3. Auflage. Springer-Verlag, New York 2006, ISBN 0-387-25494-3.
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c d e Michael T. Madigan, John M. Martinko: Brock Mikrobiologie. 11. Auflage. Addison-Wesley Verlag, München 2006, ISBN 3-8273-7187-2.
- ↑ a b Paul Vos, George Garrity, Dorothy Jones, Noel R. Krieg, Wolfgang Ludwig, Fred A. Rainey, Karl-Heinz Schleifer, William B. Whitman: Bergey’s Manual of Systematic Bacteriology: Volume 3: The Firmicutes. Springer, 2009, ISBN 978-0-387-95041-9.
- ↑ a b c d e f g h i j k Dieter Claus, Dagmar Fritze, Miloslav Kocur: Genera Related to the Genus Bacillus – Sporolactobacillus, Sporosarcina, Planococcus, Filibacter and Caryophanon (Chapter 1.2.19). In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria. 3. Auflage. Volume 4: Bacteria: Firmicutes, Cyanobacteria. Springer-Verlag, New York 2006, ISBN 0-387-25494-3, S. 631–641, doi:10.1007/0-387-30744-3_19.
- ↑ a b Georg Fuchs Thomas Eitinger, Erwin Schneider; Begründet von Hans. G. Schlegel (Hrsg.): Allgemeine Mikrobiologie. Thieme, 2007, ISBN 978-3-13-444608-1.
- ↑ a b G. Mörsdorf, H. Kaltwasser: Ammonium assimilation in Proteus vulgaris, Bacillus pasteurii, and Sporosarcina ureae. In: Archives of microbiology. Band 152, Nummer 2, 1989, S. 125–131, ISSN 0302-8933. PMID 2570557.
- ↑ a b c Sporosarcina ureae DSM 2281. In: Webseite Genomes Online Database (GOLD). Abgerufen am 11. Januar 2014.
- ↑ a b c Jean Euzéby, Aidan C. Parte: Genus Sporosarcina. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 30. Dezember 2013.
- ↑ Sporosarcina ureae DSM 2281. In: Webseite BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 11. Januar 2014.
- ↑ TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 208, abgerufen am 7. Januar 2014.
- ↑ Taxonomy Browser Sporosarcina ureae. In: Webseite des National Center for Biotechnology Information (NCBI). Abgerufen am 11. Januar 2014.
- ↑ J. A. Farrow, C. Ash, S. Wallbanks, M. D. Collins: Phylogenetic analysis of the genera Planococcus, Marinococcus and Sporosarcina and their relationships to members of the genus Bacillus. In: FEMS microbiology letters. Band 72, Nummer 2, Juni 1992, S. 167–172, ISSN 0378-1097. PMID 1505740.