Tryptophan-Synthase
| Tryptophan-Synthase | ||
|---|---|---|
| Andere Namen |
L-Serin-Hydro-Lyase | |
| Sekundär- bis Quartärstruktur | Heterotetramer | |
| Kofaktor | Pyridoxalphosphat | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 4.2.1.20, Synthase | |
| Substrat | L-Serin + 1C-(Indol-3-yl)glycerinaldehyd-3-phosphat | |
| Produkte | L-Tryptophan + D-Glycerinaldehyd-3-phosphat + H2O | |
| Vorkommen | ||
| Homologie-Familie | Hovergen | |
| Übergeordnetes Taxon | Lebewesen | |
| Ausnahmen | Animalia | |
Die Tryptophan-Synthase ist ein Enzym, das die finalen Schritte der Biosynthese von Tryptophan katalysiert.[1][2] Es kann gewöhnlich in Bakterien[3], Archaeen[4], Protisten[5], Pilzen[6] und Pflanzen[7], jedoch nicht in Tieren[8] gefunden werden. Das Enzym liegt meist als α2β2 vor.[9][10] Je eine der α-Untereinheiten katalysiert dabei die Biosynthese von Indol und dem Glycolyse-Intermediat Glycerinaldehyd-3-phosphat(GAP) aus Indol-3-glycerinphosphat (IGP). Die β-Untereinheiten hingegen ermöglichen die irreversible Kondensation von Indol und Serin zu Tryptophan. Diese Kondensationsreaktion ist vom Cofaktor Pyridoxalphosphat (PLP) abhängig. Je eine α- und eine β-Untereinheit sind über einen hydrophoben, 25 Ångström langen Kanal miteinander verbunden, was zu einer Substratkanalisierung führt.[11] Die Tryptophan-Synthase ist allosterisch reguliert.[12]
Enzymstruktur[Bearbeiten | Quelltext bearbeiten]
Untereinheiten: Für die Tryptophan-Synthase ist eine α-ββ-α-Quartärstruktur charakteristisch. Die Masse der α-Untereinheit beträgt ca. 27 kDa, während die β-Untereinheit mit 43 kDa eine etwas höhere Masse aufweist. Insgesamt besitzt das Enzym damit eine Größe von etwa 140 kDa. Die kleinere Untereinheit verfügt über eine TIM-Barrel-Struktur, während die größere Untereinheit ein Typ II-Motiv sowie eine Bindungsstelle für monovalente Kationen aufweist.[13] Die Zusammensetzung des Komplexes führt zu Strukturänderungen beider Untereinheiten und damit zur reziproken Aktivierung. Für diese Interaktion werden zwei verschiedene Mechanismen angenommen. Einerseits kann die COMM-Domäne der β-Untereinheit mit der α-Schleife der α-Untereinheit wechselwirken. Andererseits findet eine Interaktion zwischen αGly181 und βSer178 statt.[14] Die aktiven Zentren sind allosterisch reguliert und variieren so zwischen einem offenen und inaktiven, sowie einem geschlossenen, aktiven Zustand.[12]
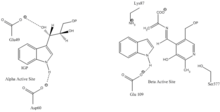
Indol-3-glycerinphosphat-Bindungsstelle: Es wird vermutet, dass die Reste αGlu49 und αAsp60 eine Rolle in der Stabilisierung des Substrates spielen (Siehe Abbildung 1).
Indol- und Serin-Bindungsstelle: In der Bindungsstelle für Indol resp. Serin scheint der Rest βGlu109 der Substratstabilisierung zu dienen. Die Reste βLys87 und βSer337 halten den Cofaktor in Position (Siehe Abbildung 1).
Hydrophober Kanal: Die α- und β-Untereinheiten sind durch einen 25 Å langen, hydrophoben Kanal miteinander verbunden, so dass das naszierende Indol von einem in das andere aktive Zentrum diffundieren kann. Diese Kanalisierung ist essentiell für die Tryptophan-Biosynthese, da das hydrophobe Indol sonst aus der Zelle diffundieren könnte.[15]
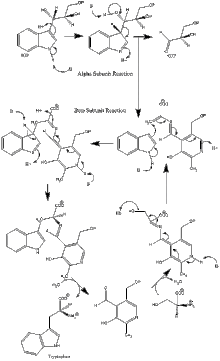
Katalytischer Mechanismus[Bearbeiten | Quelltext bearbeiten]
Reaktion der α-Untereinheit: Die α-Untereinheit katalysiert die Bildung von Indol und Glycerinaldehyd-3-phosphat durch Aldolspaltung von Indol-3-glycerinphosphat. Die Aminosäuren.αGlu49 und αAsp60 sind wahrscheinlich die katalytisch wirksamen Reste (Abbildung 1).[11] Der geschwindigkeitsbestimmende Schritt ist die Isomerisierung von IGP (Abbildung 2).[16]
Reaktion der β-Untereinheit: Die β-Untereinheit katalysiert die PLP-abhängige β-Substitution, bei der Indol und Serin miteinander zu Tryptophan und Wasser kondensieren. Die Reste βLys87, βGlu109 und βSer377 scheinen in den katalytischen Mechanismus involviert zu sein.[11] Der Mechanismus wurde jedoch noch nicht mit Sicherheit aufgeklärt. (Siehe Abbildung 2).
Nettoreaktion: (Siehe Abbildung 3).

Biologische Funktion[Bearbeiten | Quelltext bearbeiten]
Die Tryptophan-Synthase kommt in Bakterien, Archaeen, Protisten, Pilzen und Pflanzen vor, fehlt jedoch in Tieren, so auch im Menschen. Das durch die Synthase synthetisierte Tryptophan ist eine der 20 proteinogenen Aminosäuren. Da das Enzym im Menschen nicht vorkommt, zählt Tryptophan auch zu den neun essentiellen Aminosäuren und muss durch Nahrung aufgenommen werden.
Substrate[Bearbeiten | Quelltext bearbeiten]
Die Tryptophan-Synthase kann Indol-Analoga, wie beispielsweise fluorinierte oder methylierte Indolderivate, als Substrate verwenden und so zur Biosynthese entsprechender Tryptophan-Analoga führen.[17]
Medizinische Bedeutung[Bearbeiten | Quelltext bearbeiten]
Da Menschen keine eigenen Tryptophan-Synthase aufweisen, gilt sie als potentielles Target für die Medikamentenentwicklung.[18] Dieser Ansatz kann jedoch durch die hohe Diversität der Biosynthesewege von Aminosäuren unterminiert werden. Eine abschwächende Wirkung könnte allerdings ausreichen, um ein anfälliges Bakterium in einer hostilen Umgebung abzutöten. Der Tryptophan-Synthase kommt somit, wie auch anderen PLP-abhängigen Enzymen des Aminosäurestoffwechsels, Bedeutung bei der Lösung medizinischer Probleme zu.[19]
Die Inhibition der Tryptophan-Synthase und anderer PLP-Enzyme des Aminosäuremetabolismus wurde bereits für die Behandlungen folgender Krankheiten vorgeschlagen:
- Behandlung von Tuberkulose[18]
- Behandlung von Augen- und Genital-Infektionen[20]
- Behandlung von Kryptosporidiose[18]
Auch wird über einen Nutzen als Herbizid diskutiert.[21]
Evolution[Bearbeiten | Quelltext bearbeiten]
Es wird davon ausgegangen, dass das trpB2-gen bereits früh in der Evolution dupliziert wurde. Eine Kopie des Gens liegt im Tryptophan-Operon als trpB2i vor, wo zusammen es mit trpA exprimiert wird. TrpB2i formte so kurzlebige Komplexe mit TrpA, wobei es letzteres unidirektional aktiviert. Die andere Kopie des Gens verblieb außerhalb des trp-Operons als trpB2o und erfüllt eine wichtige Rolle in der Rückgewinnung von Indol aus dem Proteinabbau. TrpB2i evolvierte später zu trpB1, welches feste Komplexe mit trpA bilden kann und so zu einer bidirektionalen Aktivierung des Enzyms führt. Der Selektionsvorteil von alleinigem trpB nahm ab, so dass das solitäre Gen im Lauf der Evolution verloren ging. Schließlich fusionierten die Gene TrpB1 und TrpA, was in der Bildung des bifunktionalen Enzyms resultierte.[22]
Historische Bedeutung[Bearbeiten | Quelltext bearbeiten]
Die Tryptophan-Synthase war das erste Enzym dessen Bifunktionalität entdeckt und intensiv untersucht wurde. Außerdem war es das zuerst entdeckte Enzym, das eine Substratkanalisierung nutzt. Das Enzym ist somit von großem Interesse.[11]
Siehe auch[Bearbeiten | Quelltext bearbeiten]
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ Michael F. Dunn, Dimitri Niks, Huu Ngo, Thomas R. M. Barends, Ilme Schlichting: Tryptophan synthase: the workings of a channeling nanomachine. In: Trends in Biochemical Sciences. Band 33, Nr. 6, 1. Juni 2008, ISSN 0968-0004, S. 254–264, doi:10.1016/j.tibs.2008.04.008, PMID 18486479 (cell.com [abgerufen am 3. Dezember 2022]).
- ↑ Edith Wilson Miles: Structural Basis for Catalysis by Tryptophan Synthase. In: Advances in Enzymology - and Related Areas of Molecular Biology. John Wiley & Sons, Inc., Hoboken, NJ, USA 2006, ISBN 978-0-470-12310-2, S. 93–172, doi:10.1002/9780470123102.ch3 (wiley.com [abgerufen am 3. Dezember 2022]).
- ↑ Peter E. Jablonski, Lynn M. Jablonski, Oscar Pintado, Namalamar Sriranganathan, Carolyn J.YR 1996 Hovde: Identification of Pasteurella multocida tryptophan synthase β-subunit by antisera against strain P1059. In: Microbiology. Band 142, Nr. 1, ISSN 1465-2080, S. 115–121, doi:10.1099/13500872-142-1-115 (microbiologyresearch.org [abgerufen am 3. Dezember 2022]).
- ↑ A. Lazcano, E. Díaz-Villagómez, T. Mills, J. Oró: On the levels of enzymatic substrate specificity: Implications for the early evolution of metabolic pathways. In: Advances in Space Research. Band 15, Nr. 3, März 1995, S. 345–356, doi:10.1016/S0273-1177(99)80106-9 (elsevier.com [abgerufen am 3. Dezember 2022]).
- ↑ I Anderson: Gene Discovery in the Genome. In: Protist. Band 156, Nr. 2, 18. August 2005, S. 203–214, doi:10.1016/j.protis.2005.04.001 (elsevier.com [abgerufen am 3. Dezember 2022]).
- ↑ C. Ireland, N. Peekhaus, P. Lu, R. Sangari, A. Zhang, P. Masurekar, Z. An: The tryptophan synthetase gene TRP1 of Nodulisporium sp.: molecular characterization and its relation to nodulisporic acid A production. In: Applied Microbiology and Biotechnology. Band 79, Nr. 3, Juni 2008, ISSN 0175-7598, S. 451–459, doi:10.1007/s00253-008-1440-3 (springer.com [abgerufen am 3. Dezember 2022]).
- ↑ Sanjaya, Pao-Yuan Hsiao, Ruey-Chih Su, Swee-Suak Ko, Chii-Gong Tong, Ray-Yu Yang, Ming-Tsair Chan: Overexpression of Arabidopsis thaliana tryptophan synthase beta 1 ( AtTSB 1) in Arabidopsis and tomato confers tolerance to cadmium stress. In: Plant, Cell & Environment. Band 31, Nr. 8, August 2008, S. 1074–1085, doi:10.1111/j.1365-3040.2008.01819.x (wiley.com [abgerufen am 3. Dezember 2022]).
- ↑ S. E. Eckert, E. Kübler, B. Hoffmann, G. H. Braus: The tryptophan synthase-encoding trpB gene of Aspergillus nidulans is regulated by the cross-pathway control system. In: Molecular and General Genetics MGG. Band 263, Nr. 5, Juni 2000, ISSN 0026-8925, S. 867–876, doi:10.1007/s004380000250 (springer.com [abgerufen am 3. Dezember 2022]).
- ↑ S A Ahmed, E W Miles, D R Davies: Crystallization and preliminary X-ray crystallographic data of the tryptophan synthase alpha 2 beta 2 complex from Salmonella typhimurium. In: Journal of Biological Chemistry. Band 260, Nr. 6, März 1985, S. 3716–3718, doi:10.1016/S0021-9258(19)83682-7 (elsevier.com [abgerufen am 3. Dezember 2022]).
- ↑ C C Hyde, S A Ahmed, E A Padlan, E W Miles, D R Davies: Three-dimensional structure of the tryptophan synthase alpha 2 beta 2 multienzyme complex from Salmonella typhimurium. In: Journal of Biological Chemistry. Band 263, Nr. 33, November 1988, S. 17857–17871, doi:10.1016/S0021-9258(19)77913-7 (elsevier.com [abgerufen am 3. Dezember 2022]).
- ↑ a b c d Samanta Raboni, Stefano Bettati, Andrea Mozzarelli: Tryptophan synthase: a mine for enzymologists. In: Cellular and Molecular Life Sciences. Band 66, Nr. 14, Juli 2009, ISSN 1420-682X, S. 2391–2403, doi:10.1007/s00018-009-0028-0 (springer.com [abgerufen am 3. Dezember 2022]).
- ↑ a b M. Qaiser Fatmi, Rizi Ai, Chia-en A. Chang: Synergistic Regulation and Ligand-Induced Conformational Changes of Tryptophan Synthase. In: Biochemistry. Band 48, Nr. 41, 20. Oktober 2009, ISSN 0006-2960, S. 9921–9931, doi:10.1021/bi901358j (acs.org [abgerufen am 3. Dezember 2022]).
- ↑ Nick V. Grishin, Margaret A. Phillips, Elizabeth J. Goldsmith: Modeling of the spatial structure of eukaryotic ornithine decarboxylases. In: Protein Science. Band 4, Nr. 7, Juli 1995, S. 1291–1304, doi:10.1002/pro.5560040705, PMID 7670372, PMC 2143167 (freier Volltext) – (wiley.com [abgerufen am 3. Dezember 2022]).
- ↑ Thomas R. Schneider, Eva Gerhardt, Minsu Lee, Po-Huang Liang, Karen S. Anderson, Ilme Schlichting: Loop Closure and Intersubunit Communication in Tryptophan Synthase ,. In: Biochemistry. Band 37, Nr. 16, 21. April 1998, ISSN 0006-2960, S. 5394–5406, doi:10.1021/bi9728957 (acs.org [abgerufen am 3. Dezember 2022]).
- ↑ Xinyi Huang, Hazel M. Holden, Frank M. Raushel: Channeling of Substrates and Intermediates in Enzyme-Catalyzed Reactions. In: Annual Review of Biochemistry. Band 70, Nr. 1, Juni 2001, ISSN 0066-4154, S. 149–180, doi:10.1146/annurev.biochem.70.1.149 (annualreviews.org [abgerufen am 3. Dezember 2022]).
- ↑ K. S. Anderson, E. W. Miles, K. A. Johnson: Serine modulates substrate channeling in tryptophan synthase. A novel intersubunit triggering mechanism. In: The Journal of Biological Chemistry. Band 266, Nr. 13, 5. Mai 1991, ISSN 0021-9258, S. 8020–8033, PMID 1902468.
- ↑ Michael Wilcox: The enzymatic synthesis of l-tryptophan analogues. In: Analytical Biochemistry. Band 59, Nr. 2, Juni 1974, S. 436–440, doi:10.1016/0003-2697(74)90296-6 (elsevier.com [abgerufen am 4. Dezember 2022]).
- ↑ a b c Kshitiz Chaudhary, David S Roos: Protozoan genomics for drug discovery. In: Nature Biotechnology. Band 23, Nr. 9, September 2005, ISSN 1087-0156, S. 1089–1091, doi:10.1038/nbt0905-1089, PMID 16151400, PMC 7096809 (freier Volltext) – (nature.com [abgerufen am 4. Dezember 2022]).
- ↑ Daniel Becker, Matthias Selbach, Claudia Rollenhagen, Matthias Ballmaier, Thomas F. Meyer, Matthias Mann, Dirk Bumann: Robust Salmonella metabolism limits possibilities for new antimicrobials. In: Nature. Band 440, Nr. 7082, März 2006, ISSN 0028-0836, S. 303–307, doi:10.1038/nature04616 (nature.com [abgerufen am 4. Dezember 2022]).
- ↑ Harlan D. Caldwell, Heidi Wood, Debbie Crane, Robin Bailey, Robert B. Jones, David Mabey, Ian Maclean, Zeena Mohammed, Rosanna Peeling, Christine Roshick, Julius Schachter, Anthony W. Solomon, Walter E. Stamm, Robert J. Suchland, Lacey Taylor, Sheila K. West, Tom C. Quinn, Robert J. Belland, Grant McClarty: Polymorphisms in Chlamydia trachomatis tryptophan synthase genes differentiate between genital and ocular isolates. In: Journal of Clinical Investigation. Band 111, Nr. 11, 1. Juni 2003, ISSN 0021-9738, S. 1757–1769, doi:10.1172/JCI17993, PMID 12782678, PMC 156111 (freier Volltext) – (jci.org [abgerufen am 4. Dezember 2022]).
- ↑ Victor Kulik, Elisabeth Hartmann, Michael Weyand, Monika Frey, Alfons Gierl, Dimitri Niks, Michael F. Dunn, Ilme Schlichting: On the Structural Basis of the Catalytic Mechanism and the Regulation of the Alpha Subunit of Tryptophan Synthase from Salmonella typhimurium and BX1 from Maize, Two Evolutionarily Related Enzymes. In: Journal of Molecular Biology. Band 352, Nr. 3, September 2005, S. 608–620, doi:10.1016/j.jmb.2005.07.014 (elsevier.com [abgerufen am 4. Dezember 2022]).
- ↑ Sonja Leopoldseder, Stefan Hettwer, Reinhard Sterner: Evolution of Multi-Enzyme Complexes: The Case of Tryptophan Synthase. In: Biochemistry. Band 45, Nr. 47, 1. November 2006, ISSN 0006-2960, S. 14111–14119, doi:10.1021/bi061684b (acs.org [abgerufen am 4. Dezember 2022]).
